プレスリリース 「新たな研究手法開発によりがん抑制microRNA-34aの標的遺伝子を同定」―microRNA-34aによる乳がん抑制機能に重要な標的遺伝子を同定―
プレスリリース
国立大学法人東京医科歯科大学
国立研究開発法人日本医療研究開発機構
ポイント
- がんなどの疾患に関わるmicroRNAの新たな標的遺伝子の同定法を開発しました。
- 本手法により、がん抑制microRNA-34aの標的遺伝子として、乳がん増悪化に関わるGFRA3を同定しました。
東京医科歯科大学大学院医歯学総合研究科システム発生・再生医学分野の淺原弘嗣教授、伊藤義晃プロジェクト助教の研究グループは、国立成育医療研究センター、名古屋市立大学、インペリアル・カレッジ・ロンドン、カリフォルニア工科大学およびベックマン研究所との共同研究で、がんなど様々な疾患において重要な働きを担うmicroRNAの標的遺伝子を同定する新たな方法を開発し、がん抑制microRNA-34aの新たな標的遺伝子として、乳がんの増悪化に関わるGFRA3を同定しました。この研究は文部科学省科学研究費補助金、国立研究開発法人日本医療研究開発機構(AMED)の革新的先端研究開発支援事業(AMED-CREST)「炎症の慢性化機構の解明と制御に向けた基盤技術の創出」研究開発領域(研究開発総括:宮坂昌之)における研究開発課題「RNA階層における炎症の時間軸制御機構の解明」(研究開発代表者:淺原弘嗣)ならびに米国国立衛生研究所(NIH,NIAMS)の支援のもとで行われました。なお、AMED-CRESTの研究開発領域は、平成27年4月の日本医療研究開発機構の発足に伴い、国立研究開発法人科学技術振興機構(JST)より移管されたものです。この研究成果は、国際科学誌Proceedings of the National Academy of Sciences USA(米国科学アカデミー紀要)に、2017年3月27日(米国時間)の週に発表されます(文献)。
研究の背景
研究成果の概要
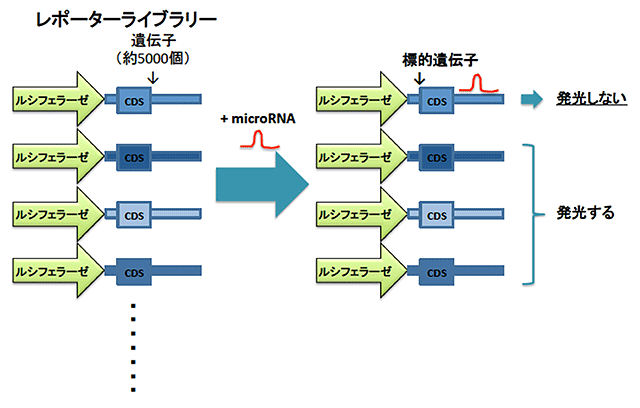 【図1】レポーターライブラリーシステム
【図1】レポーターライブラリーシステム
ルシフェラーゼ遺伝子の3’UTRに約5000遺伝子の全長cDNAが含まれるレポーターライブラリーとmicroRNAを細胞内に導入し、ルシフェラーゼ活性の低下の有無によりmicroRNAの標的遺伝子であるかを判定するシステムを開発した。
本手法を用いてがん抑制microRNAであるmiR-34aの標的遺伝子の探索を行いました。その結果、既に報告されている標的遺伝子に加え、新規な標的遺伝子として、GFRA3、FAM76A、REM2およびCARKLを同定しました(図2)。また、microRNAは主に標的遺伝子の3’UTRを介して制御すると言われていますが、GFRA3はタンパク質をコードしているコーディング領域(CDS)を介して直接制御されていることが明らかになりました(図3)。
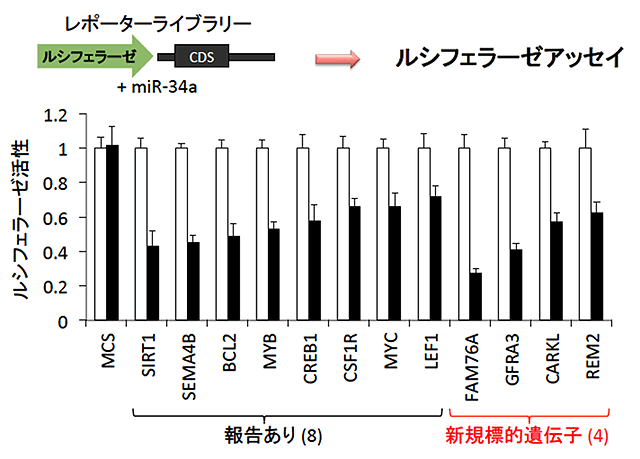 【図2】レポーターライブラリーシステムによるmiR-34a標的遺伝子の同定
【図2】レポーターライブラリーシステムによるmiR-34a標的遺伝子の同定
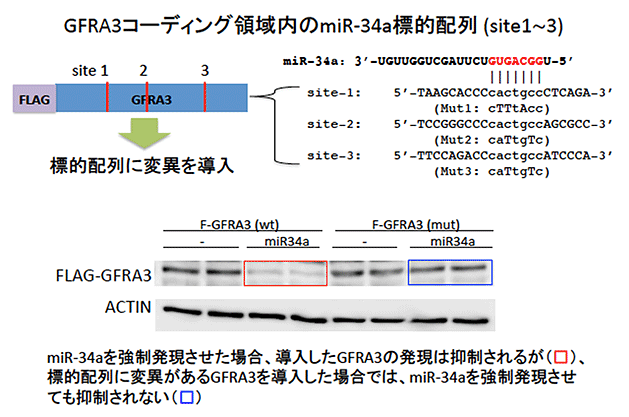 【図3】GFRA3は3’UTRを介して制御される
【図3】GFRA3は3’UTRを介して制御される
新たにmiR-34aの標的遺伝子として同定したGFRA3は、コーディング領域に標的配列があり、通常のGFRA3 (wt)ではmiR-34aによりその発現は抑制されるが(赤い四角で囲った部分)、その標的配列に変異を導入したGFRA3では (mut)、miR-34aによる抑制が見られない(青い四角で囲った部分)。
このGFRA3をMDA-MB-231という乳がん細胞においてノックダウンすると、MDA-MB-231細胞の増殖が抑制されました。またGFRA3の発現が高い乳がんを持つ患者は、GFRA3の発現が低い乳がんを持つ患者に比べて生存率が低いことが分かりました(図4)。これらの結果から、GFRA3は乳がん増悪化に関わる重要な遺伝子であることが明らかになりました。
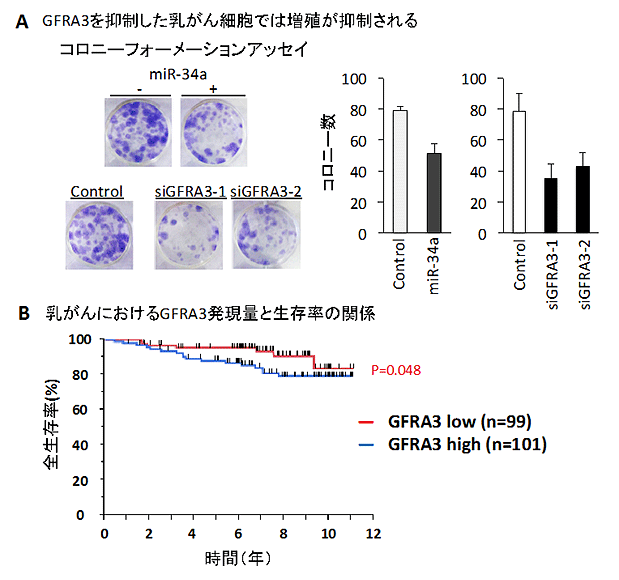 【図4】GFRA3は乳がんの増悪化因子である
【図4】GFRA3は乳がんの増悪化因子である
研究成果の意義
文献
Ito, Y., et al., Identification of targets of tumor suppressor microRNA-34a using a reporter library system. Proc Natl Acad Sci U S A, 2017. xx(x): p - .
図2から4は文献より引用、一部改変。
お問い合わせ先
研究に関すること
東京医科歯科大学大学院医歯学総合研究科
システム発生・再生医学分野
淺原 弘嗣(アサハラ ヒロシ)
伊藤 義晃(イトウ ヨシアキ)
TEL:03-5803- 5015 FAX:03-5803- 5810
E-mail:asahara.syst“AT”tmd.ac.jp
報道に関すること
東京医科歯科大学 広報部広報課
〒113-8510 東京都文京区湯島1-5-45
TEL:03-5803-5833 FAX:03-5803-0272
E-mail:kouhou.adm“AT”tmd.ac.jp
事業に関すること
国立研究開発法人日本医療研究開発機構(AMED)
戦略推進部 研究企画課
TEL:03-6870-2224 FAX:03-6870-2243
E-mail:kenkyuk-ask“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
掲載日 平成29年3月28日
最終更新日 平成29年3月28日


