ヒト・セントロメアDNA配列の隠された多様性を解明
プレスリリース
東京大学
日本医療研究開発機構
発表者
鈴木裕太(東京大学大学院新領域創成科学研究科 メディカル情報生命専攻 特任助教)
森下真一(東京大学大学院新領域創成科学研究科 メディカル情報生命専攻 教授)
発表のポイント
- さまざまな民族由来の36サンプルのゲノム情報を分析することで、ヒトゲノム中で正常な細胞分裂に関わる重要な機能領域であるセントロメアDNAの配列多様性を解明しました。
- 長鎖DNA配列決定技術と、新たに開発した解析ソフトウェアとを組み合わせることで、セントロメア領域のような複雑な繰り返し配列からなる難読領域において、大規模なDNA配列の挿入・欠失を伴った構造変異などの配列多様性を分析することができました。
- 全ゲノム関連解析(GWAS)からセントロメア周辺のDNA配列と疾患との関連が示唆されており、今後はセントロメア内部の配列多様性を調べることで、さまざまな遺伝的要因に由来する疾患などとの関係について、さらなる理解が期待できます。
発表概要
ヒトゲノム(注1)において、セントロメア(注2)はヒトの正常な細胞分裂に関わる重要な機能をもった領域です。セントロメア領域に疾患関連変異が存在する可能性が、従来の全ゲノム関連解析(Genome Wide Association Study;GWAS)では示唆されています。しかし、ヒトゲノムのセントロメア領域のDNA配列は、特殊な繰り返しの塩基配列で構成されているために、その解読が難しく、詳細な分析は困難でした。
今回、東京大学大学院新領域創成科学研究科の鈴木裕太特任助教と森下真一教授、およびマックス・プランク分子細胞生物学・遺伝学研究所(ドイツ、ドレスデン)のEugene Myers博士らの国際共同研究チームは、ヒトのセントロメア領域を長鎖DNA配列決定技術(注3)で得られたデータに基づいて解析する手法を開発し、さまざまな民族由来の36サンプル(21サンプルの日本人由来のゲノムDNAを含む)を比較・分析することで、セントロメア領域のDNA配列には大きな多様性が存在することを明らかにしました。
このようなセントロメア領域のDNA配列の多様性の意味は今のところ不明ですが、今後はさまざまな遺伝的要因に由来する疾患解明が、セントロメア領域の配列多様性と細胞機能との関係性から深く理解されることが期待されます。
本研究は、国立研究開発法人日本医療研究開発機構(AMED)ゲノム医療実現推進プラットフォーム事業(先端ゲノム研究開発:GRIFIN)の支援を受けて実施されました。
発表内容
研究の背景
セントロメアは動原体(細胞分裂時に染色体を分離する力がはたらく点)が形成される重要な機能領域です。多くの生物種で、この領域のDNAは特殊な繰り返しの塩基配列から構成されており、例えば、ヒトのセントロメア領域のDNA配列はHOR(Higher-Order Repeat、注4)という階層的な構造をしています。そこでは、およそ171塩基対の小さな単位(モノマー)が、2個から34個並ぶことで、より大きな「HOR単位」を形成し、このHOR単位が数百~数千回繰り返しています(図1)。
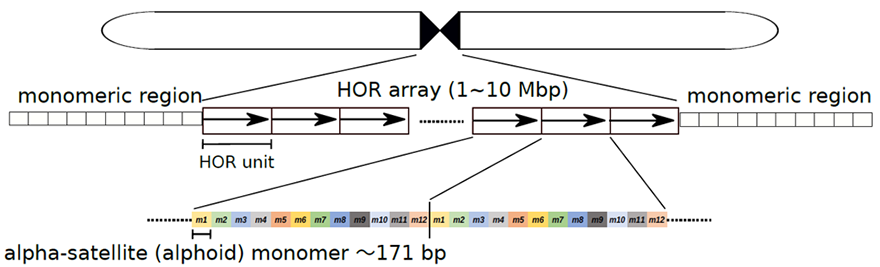
ところで、ヒト個人間の遺伝的な差異は、各々の個人がもつDNA配列の差異に基づいています。セントロメア領域においても、各々の個人がもつDNA配列に差異があることが示唆されていました。例えば、性染色体のひとつであるX染色体のセントロメア領域の長さは個人によって差異があり、最大で10倍程度の違いが存在することが分かっています。また、セントロメア領域を構成するHORには、染色体ごとに決まった典型的な組み合わせがありますが、大規模なDNA配列の挿入や欠失を伴った構造変異(注5)を含む、低頻度の変異HORの存在も知られており、このような変異HORが個人ごとにどのように分布しているのかは、従来の研究手法で調べることは困難でした。実際、セントロメア領域のもつ特殊な繰り返しの塩基配列のために、最新の長鎖DNA配列決定技術を駆使しても、正確な配列決定は容易ではないと考えられています。したがって、セントロメア領域のDNA配列の多様性の全貌については、よく分かっていませんでした。
研究手法と成果
今回、東京大学大学院新領域創成科学研究科の鈴木裕太特任助教と森下真一教授、およびマックス・プランク分子細胞生物学・遺伝学研究所(ドイツ、ドレスデン)のEugene W. Myers博士の国際共同研究チームは、長鎖DNA配列決定技術で得られたデータをもとに、セントロメア領域におけるDNA配列の多様性を解析するための新たな計算手法を開発しました。この手法では、データから得られたDNA断片配列上でまず基本となる単位(モノマー)を検出し、引き続いてHORに対応する領域をモノマーの組合せとして検出することで、解析サンプルごと、DNA配列中に存在する「高頻度の典型的HOR単位」、および「低頻度の変異HOR単位」を網羅的に同定することができます(図2)。
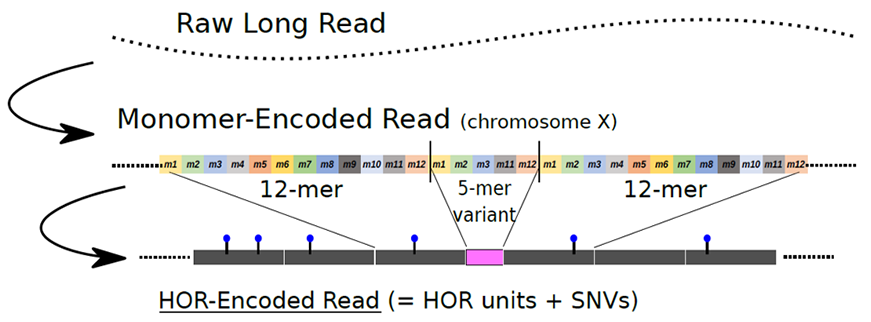
本共同研究チームはこの手法を用いて、長鎖DNA配列決定技術によって得られた21サンプルの日本人のDNA配列データと、同様のデータが公開データベース上で利用可能な(アフリカ・ヨーロッパ・アジア・アメリカの各地域の個人に由来する)15サンプルのDNA配列データから、計36サンプルのセントロメア領域のDNA配列データを分析・比較しました。その結果、「低頻度の変異HOR」の分布に、個人間でセントロメア領域のDNA配列の多様性が大きく存在していることが分かりました。
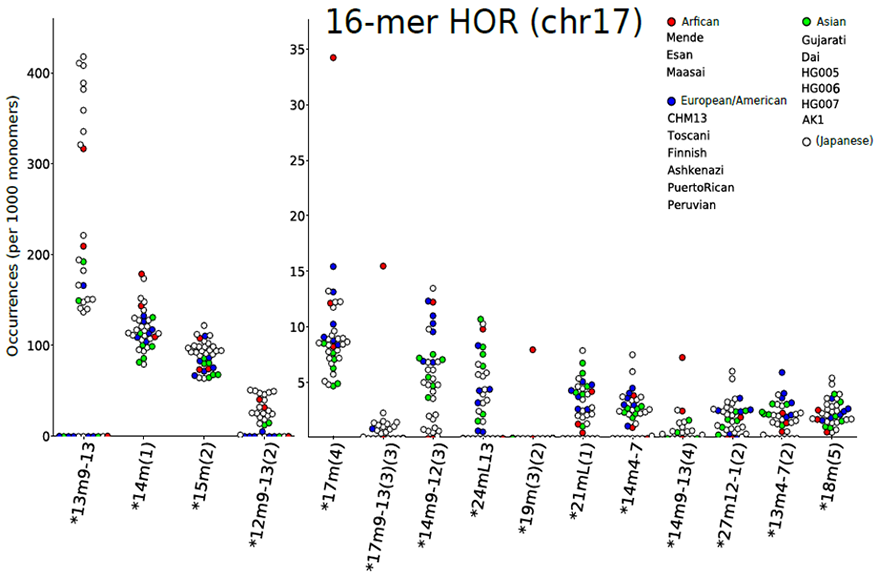
例えば、17番染色体のセントロメア領域は16個のモノマーからなる高頻度のHOR単位から構成されています。今回、23サンプルのセントロメア領域のDNA配列データを分析・比較したところ、13個のモノマーからなる「低頻度の変異HOR」が多数検出されました(図3)。この変異HORの存在自体はすでに報告されているものでしたが、今回の網羅的解析によって、さらに多くの種類の変異HORの存在を明らかにすることができました。
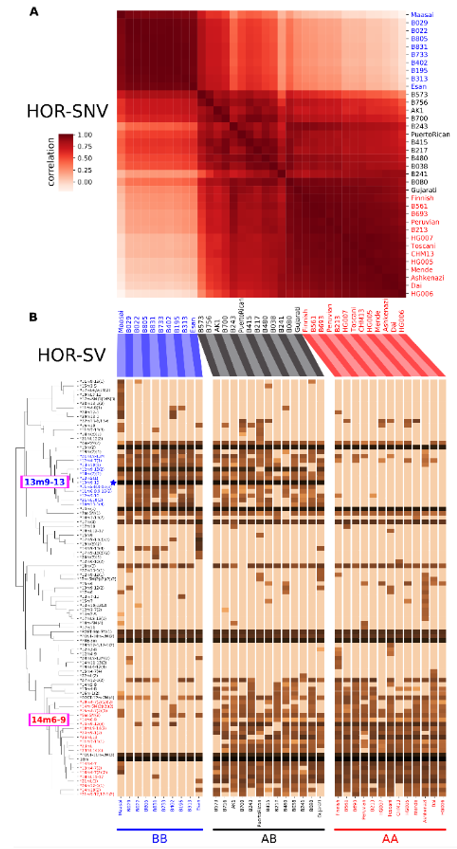
特に、変異HORの分布パターンにより17番染色体のセントロメア領域は、2種類のパターン(アレルAまたはアレルB)に分類できる様子が明瞭になり、各個人のDNA配列はこの2種類のアレルの組み合わせから、AAのホモ接合(同一のアレルの組合せ)とBBのホモ接合、ABのヘテロ接合(別種のアレルの組合せ)の3通りの状態に分類されました。このことは、17番染色体のセントロメア全体が単一の遺伝子座(遺伝情報のひとかたまり)としてふるまっていることを示し、「セントロメア領域においては遺伝的組み換えが起こりにくい」という仮説を支持する結果となりました(図4)。
今後期待される展開
セントロメア領域における特殊な繰返しの塩基配列構造は、全く同一の「高頻度のHOR単位」の繰り返しからなるのではなく、「低頻度の変異HOR」を多数含んでいるという事実は、現在みられるセントロメアの配列構造がどのような機序で進化してきたのか、その履歴を復元できる可能性を示唆しています(図5)。
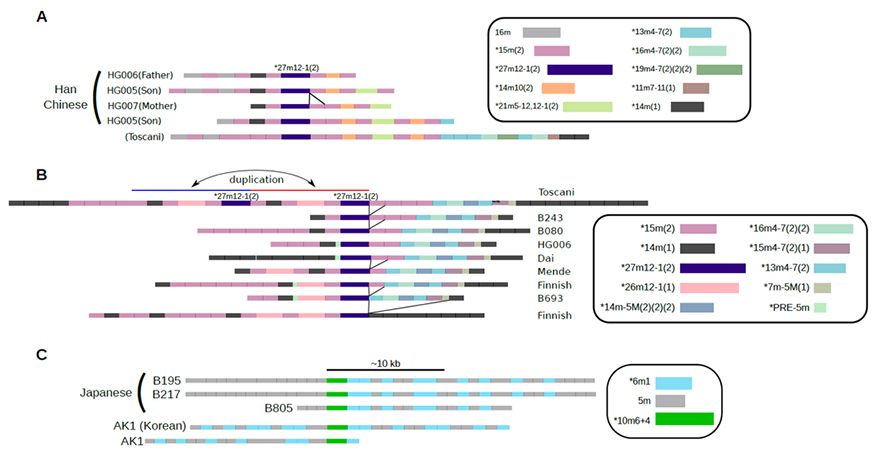
各サンプルの長鎖DNA配列から、高度に類似したパターンを並べたもの。上段は、両親(HG006、HG007)と子(HG005)の間でパターンが引き継がれる例にもなっている。
また、今回解明された「セントロメア領域のDNA配列の多様性が非常に大きい」という事実は、この領域の機能的な意義や、ヒトの個体差への寄与を再検討する必要を示唆しています。例えば、セントロメア領域の周縁には、さまざまな遺伝的要因に由来する疾患に相関する遺伝的多型が多数検出されることが知られています。それ故、このような疾患の真の原因がセントロメア領域の配列構造の違いにある可能性が高まり、その可能性を臨床的な視点と結びつけるための意義への貢献が期待されます。
発表雑誌
- 雑誌名
- 「Science Advances」(オンライン版:2020年12月11日)
- 論文タイトル
- Rapid and ongoing evolution of repetitive sequence structures in human centromeres
- 著者
- Yuta Suzuki, Eugene W. Myers, Shinichi Morishita
- DOI
- 10.1126/sciadv.abd9230
用語解説
- (注1)ゲノム
- 個体がもつ遺伝情報の総体を指す。その本体はDNAという生体高分子であり、遺伝情報は4種の塩基(A、C、G、T)からなる配列に保持されている。全染色体を構成するDNAの全塩基配列の個体間の違いが遺伝的多様性を生み出している。
- (注2)セントロメア
- 細胞分裂の際に、一時的に凝縮される染色体(凝縮染色体)の形成に重要な機能を果たし、染色体上ではくびれた形態を示す。非常にゲノムDNAの密度が高い領域であり、特殊な繰返しの塩基配列構造のため、塩基配列決定が難しい。
- (注3)長鎖DNA配列決定技術
- 2010年代より実用化された新しい塩基配列決定手法で、いわゆる次世代シークエンシング法と呼ばれる従来型と比較して、より長い配列(数千塩基対から数万塩基対以上のいわゆるロングリード)を同時に決定することができる。第3世代シークエンシングとも呼ばれる。
- (注4)HOR(Higher-Order Repeat)
- ヒトや一部の霊長類のセントロメア領域に見られる、階層性をもった特殊な繰返しの塩基配列構造。
- (注5)構造変異
- ゲノムDNA上の塩基配列における差異のうち、数十塩基対から数万塩基対以上の増減を伴う変化を指す。長鎖DNA配列決定技術の登場により検出が容易になった。
お問い合わせ先
研究に関すること
東京大学大学院新領域創成科学研究科 メディカル情報生命専攻
特任助教 鈴木裕太(すずきゆうた)
Tel:04-7136-3985
E-mail:yuta_suzuki“AT”edu.k.u-tokyo.ac.jp
報道に関すること
東京大学大学院新領域創成科学研究科 広報室
Tel:04-7136-5450
E-mail:press“AT”k.u-tokyo.ac.jp
AMED事業に関すること
日本医療研究開発機構(AMED)
ゲノム・データ基盤事業部 ゲノム医療基盤研究開発課
ゲノム医療実現推進プラットフォーム事業(先端ゲノム研究:GRIFIN)担当
Tel:03-6870-2228
E-mail:genome-platform“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
関連リンク
掲載日 令和2年12月14日
最終更新日 令和2年12月14日


