プレスリリース 白質消失病の発症機構―大脳白質「消失」の鍵を握る巨大タンパク質の立体構造を解明―
プレスリリース
国立大学法人東京大学
国立研究開発法人日本医療研究開発機構
要旨
理化学研究所(理研)横山構造生物学研究室の横山茂之上席研究員(東京大学名誉教授)と、理研ライフサイエンス技術基盤研究センター翻訳因子構造解析研究ユニットの伊藤拓宏ユニットリーダー、柏木一宏特別研究員らの研究グループ※は、白質消失病発症の原因タンパク質「eIF2B」の立体構造を結晶構造解析により解明し、分子レベルでeIF2Bのストレス応答機構を明らかにしました。
白質消失病は幼児期に発症し、ウイルス感染や頭部外傷などのストレスを契機に急速に悪化し、大脳の白質が消失して運動機能の失調をきたす遺伝性の神経変性疾患です。罹患者の多くが死に至る深刻な疾患で、この疾患の原因となる翻訳開始因子[1]eIF2Bは、本来、細胞がタンパク質を合成する際に他の翻訳開始因子eIF2を活性化するタンパク質です。細胞がストレスを受けると、eIF2がリン酸化してeIF2Bの活性が低下し、一般的なタンパク質合成がいったん抑制されます。しかし、このストレス応答機構や、白質消失病の発症との関連性は解明されておらず、白質消失病の治療法はありません。
研究グループは、大型放射光施設「SPring-8[2]」を用いたX線結晶構造解析で、10個のサブユニットから構成される巨大なeIF2Bの3次元構造を解明しました。その結果、eIF2Bの変異の大半は、eIF2に働く領域(活性部位)や、サブユニット間の相互作用面に集中していることが分かりました。これは、変異によって活性部位や全体構造が損なわれ、eIF2Bの機能が低下することが、白質消失病の発症原因であることを意味します。ストレスを受けて一般的なタンパク質合成が抑制された後、抑制が解除されてストレス状態から通常状態へと回復する際に、eIF2Bの機能が低下しているために回復に必要なタンパク質を十分に合成できず、神経細胞が変性し白質消失に至ると考えられます。さらに、独自開発した技術を活用しeIF2BとeIF2に非天然型アミノ酸を導入して解析したところ、eIF2Bの3次元構造の上でリン酸化されたeIF2と結合する領域は、通常のeIF2を活性化する際に結合する領域とは異なることを発見しました。この結合様式によりリン酸化されたeIF2は、自身の活性化を起こさないだけでなく、他のeIF2の活性化も妨げることが分かりました。
これらの成果は、白質消失病などの病態の理解、eIF2Bを標的としたストレス応答を制御する治療法の開発へ向けて、有用な基礎的情報となると期待できます。
本研究は、文部科学省ターゲットタンパク研究プログラム、文部科学省及び国立研究開発法人日本医療研究開発機構 創薬等ライフサイエンス研究支援基盤事業(創薬等支援技術基盤プラットフォーム事業)、JSPS科研費と東京大学大学院理学系研究科構造生物学社会連携講座の支援により行われました。成果は、英国の科学雑誌『Nature』(3月3日号)に掲載されるのに先立ち、オンライン版(2月22日付け:日本時間2月23日)に掲載されます。
背景
研究手法と成果
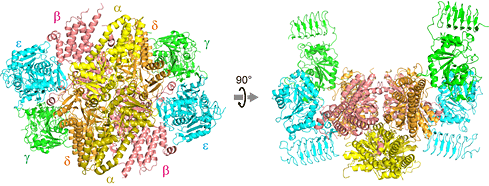 図1 eIF2Bの立体構造
図1 eIF2Bの立体構造
X線結晶構造解析によって決定された分裂酵母eIF2Bの全体構造。α、β、δサブユニットが構造の中心で六量体を形成し、γ、εサブユニットの二量体がその両側に1つずつ結合している。
続いて、独自に開発した非天然型アミノ酸導入技術を用いるSurface-scanning photo-cross-linking法[3]を使って、eIF2BがeIF2と相互作用する領域を解析しました。eIF2はα、β、γの3種類のサブユニットで構成される複合体です。eIF2のγサブユニットがeIF2Bのγεサブユニットの外側の面に、eIF2のαサブユニットがeIF2Bのαβδサブユニットで囲まれた中央部に、それぞれ結合することが明らかになりました(図2左)。GDPやGTPが結合するのはeIF2のγサブユニットであり、ストレスによってリン酸化されるのはeIF2のαサブユニットであるため、eIF2の活性化が外側の領域で、そしてリン酸化したeIF2の認識が中央部でそれぞれ行われると考えられます。
解析により明らかになった2カ所のeIF2との結合領域は離れて位置しており、1分子のeIF2が両方の領域に同時に結合することも、2分子のeIF2がそれぞれ異なる領域に同時に結合することも、いずれも困難な位置関係でした(図2右)。
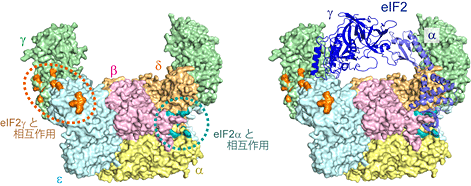 図2 eIF2BのeIF2との相互作用領域
図2 eIF2BのeIF2との相互作用領域
左:Surface-scanning photo-cross-linkingの際に、eIF2Bの構造のうちeIF2のγサブユニットと結合する位置を橙色、eIF2のαサブユニットと結合する位置を青緑色で示した。eIF2の活性化はγサブユニットへの作用であり、eIF2のリン酸化はαサブユニットに対して起こるため、橙色で囲んだ領域で活性化のための相互作用が起き、また、青緑色で囲んだ領域でリン酸化状態の識別のための相互作用が行われていると考えられる。
右:eIF2BとeIF2の複合体構造。eIF2のαサブユニットがeIF2Bの青緑色の領域に結合するようにeIF2を配置している。この結合状態をとるとき、eIF2のγサブユニットは橙色の領域まで到達できない(簡略化のためeIF2βサブユニットは描かれていない)。
また、eIF2のリン酸化によって、eIF2BとeIF2は強固な複合体を形成することが確認できました。これらから、ストレス環境下でリン酸化されたeIF2はeIF2Bの中央部に強く結合し、その結果、eIF2Bの外側の領域で行われるeIF2の活性化については、リン酸化したeIF2自身の活性化のみでなく、他のeIF2分子の活性化までもが妨げられる、という分子レベルでのストレス応答機構が明らかになりました(図3)。
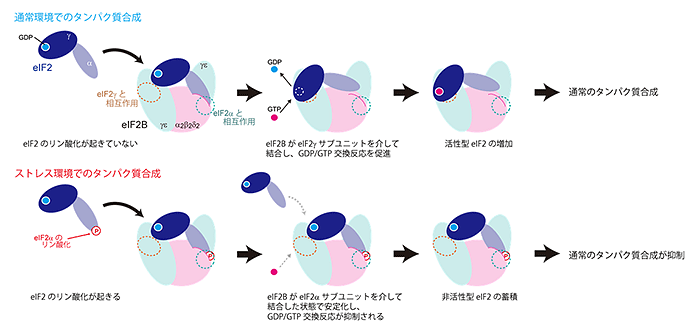 図3 eIF2Bを介したストレス応答機構
図3 eIF2Bを介したストレス応答機構
eIF2BとeIF2との相互作用には、eIF2のαサブユニットを介して結合した状態と、γサブユニットを介したeIF2の活性化に適した結合状態が存在する。通常の環境では、eIF2γのサブユニットを介した結合によってGDP/GTP交換反応が促進される(上)。一方、ストレス環境下では、eIF2のリン酸化によってeIF2のαサブユニットを介した結合状態が安定化され、GDP/GTP交換反応や他のeIF2分子の結合が妨げられる。その結果、活性型eIF2が減少して通常のタンパク質合成は抑制される(下)。
白質消失病患者にこれまでに確認されているeIF2Bの100カ所以上の変異の多くは、eIF2のγサブユニットとの相互作用領域(GDP/GTP交換に必要な部位)の周辺やeIF2Bのサブユニット間の相互作用面の近傍に位置していることが分かりました(図4)。このことは、白質消失病の発症が、遺伝子の変異により、eIF2BがeIF2と相互作用する部位や全体の構造が影響を受け、eIF2を活性化する能力が低下することと関連していることを示唆しています。
以上のことから、ストレスを受けて一時的に一般的なタンパク質合成が抑制された後、抑制が解除されてストレス状態から通常状態へと回復する際にタンパク質を十分に合成できないために神経細胞が変性し白質消失に至ると考えられます。
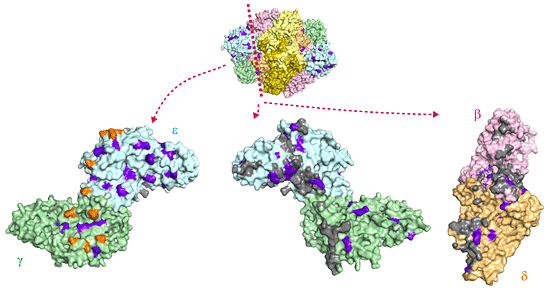 図4 白質消失病患者で確認されたeIF2Bの変異の分布
図4 白質消失病患者で確認されたeIF2Bの変異の分布
今後の期待
論文情報
タイトル
Crystal structure of eukaryotic translation initiation factor 2B
著者名
Kazuhiro Kashiwagi, Mari Takahashi, Madoka Nishimoto, Takuya B. Hiyama, Toshiaki Higo, Takashi Umehara, Kensaku Sakamoto, Takuhiro Ito and Shigeyuki Yokoyama
雑誌
Nature
DOI
10.1038/nature16991
補足説明
- [1]翻訳開始因子
- 細胞内でタンパク質の合成(翻訳)を行うリボソームが、合成を開始する際に必要とするタンパク質群。
- [2]SPring-8
- 理化学研究所が所有する、兵庫県の播磨科学公園都市にある世界最高輝度の放射光を生み出す共用施設。SPring-8の名前はSuper Photon ring-8 GeVに由来する。放射光とは、荷電粒子が磁場の中で曲がる際に放射される光の一種。SPring-8では、周回する電子群のサイズが小さいことや高い安定性のため、干渉性の優れたX線が得られる。
- [3]Surface-scanning photo-cross-linking法
- タンパク質の表面にあるアミノ酸残基が非天然型アミノ酸であるパラベンゾイルフェニルアラニン(pBpa)に置き換えられた変異体を用いて、標的タンパク質との結合表面を特定する方法。pBpaは、光によって活性化されて近くの炭素原子と共有結合を形成する性質を持つため、pBpaが結合表面の近くに位置する変異体では標的タンパク質と結合する。さまざまな位置のアミノ酸残基をpBpaに置き換えて架橋形成の有無を調べることで、相互作用が起こっている領域を明らかにできる。
お問い合わせ先
発表者 ※研究内容については発表者にお問い合わせください
理化学研究所
横山構造生物学研究室
上席研究員 横山 茂之(よこやま しげゆき)
(東京大学名誉教授)
TEL:045-503-9196(横山) FAX:045-503-9195(横山)
E-mail:yokoyama“AT”riken.jp(横山)
ライフサイエンス技術基盤研究センター 構造・合成生物学部門
構造生物学グループ 翻訳因子構造解析研究ユニット
ユニットリーダー 伊藤 拓宏(いとう たくひろ)
特別研究員 柏木 一宏(かしわぎ かずひろ)
TEL:045-503-9204(伊藤) FAX:045-503-9201(伊藤)
E-mail:takuhiro.ito“AT”riken.jp(伊藤)
機関窓口
理化学研究所 広報室 報道担当
TEL:048-467-9272 FAX:048-462-4715
E-mail:ex-press“AT”riken.jp
東京大学 大学院理学系研究科・理学部広報室
特任専門職員 武田加奈子、准教授・広報室副室長 横山広美
TEL:03-5841-8856
E-mail:kouhou“AT”adm.s.u-tokyo.ac.jp
AMED事業について
日本医療研究開発機構 戦略推進部医薬品研究課
TEL:03-6870-2219 FAX:03-6870-2244
E-mail:ddlsg“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
※研究グループ
理化学研究所
横山構造生物学研究室
上席研究員 横山 茂之(よこやま しげゆき)*
ライフサイエンス技術基盤研究センター 構造・合成生物学部門
翻訳因子構造解析研究ユニット
ユニットリーダー 伊藤 拓宏(いとう たくひろ)*
特別研究員 柏木 一宏(かしわぎ かずひろ)**
リサーチアソシエイト 高橋 真梨(たかはし まり)
技師 西本 まどか(にしもと まどか)
非天然型アミノ酸技術研究チーム
チームリーダー 坂本 健作(さかもと けんさく)
エピジェネティクス制御研究ユニット
ユニットリーダー 梅原 崇史(うめはら たかし)
元研修生 肥後 聡明(ひご としあき)
生命分子システム基盤研究領域 システム研究チーム
元特別研究員 檜山 卓也(ひやま たくや)
* 2012年3月まで、** 2014年3月まで東京大学大学院理学系研究科
掲載日 平成28年2月23日
最終更新日 平成28年2月23日


