プレスリリース 日本人の基準ゲノム配列(JRG)を決定―長鎖読みとり型次世代シークエンサーを用いて日本人のもつゲノム構造を解明―
プレスリリース
東北大学 東北メディカル・メガバンク機構
国立研究開発法人日本医療研究開発機構
発表のポイント
- 長鎖読みとり型次世代シークエンサーを用いて、全ゲノム解読を完了。従来の技術では精確な解読が困難だった約3,700箇所の挿入配列の解読に成功。
- 国際ヒトゲノム参照配列に約250万塩基を新規追加した、日本人の基準ゲノム配列(JRG)を公開へ
- 日本人の基準ゲノムを作成することで日本人ゲノム解析の精度向上と、結果として疾患関連遺伝的多様体の高精度検出・同定が期待できる。
概要
東北大学東北メディカル・メガバンク機構(以下、ToMMo)は、コホート調査*1の参加者から提供されたDNAをもとに、長鎖読みとり型の次世代シークエンサー*2PacBio RS II(Pacific Bioscience社製:以下、Pac Bio)を用いて、ヒトゲノム全長の100倍に相当する3,000億塩基のシークエンシングを行い、全ゲノム解読しました。本シークエンス解析解読の結果、国際ヒトゲノム参照配列*3に対して、日本人が保有しこれまで報告されてこなかった約3,700箇所の新たな挿入配列、約250万塩基の同定に成功しました。この解読完了から、ToMMoでは、新たに同定された配列群を挿入するなどして得られた日本人の基準ゲノム配列JRG v1(Japanese Reference Genome version 1)、を、デコイ配列*4decoyJRG v1と共に公開することとしました。デコイ配列とは、国際ヒトゲノム参照配列には含まれていない領域をまとめたもので、decoyJRG v1は日本人に高頻度でありながら国際ヒトゲノム参照配列には含まれていない配列で、ゲノム解読時の読み取り精度の向上に活用されます。
両配列の公開は、国際ヒトゲノム参照配列だけを用いている際には精確に読みとることのできなかった領域の研究に大きく寄与し、日本のゲノム研究全体を底上げ、加速させるものと期待されます。また今回の成果は日本人に特徴的なゲノム構造を明らかにする成果であり、今後、日本の医学研究の大きな基盤となる成果と考えられます。
背景
東北大学と岩手医科大学は、2012年から東日本大震災の被害からの復興事業として、東北メディカル・メガバンク計画に取り組み、東北大学は東北メディカル・メガバンク機構(以下、ToMMo)を、岩手医科大学はいわて東北メディカル・メガバンク機構をそれぞれ設立して事業を進めています。両機構は、宮城・岩手両県の住民15万人に対し健康調査(コホート調査)を実施することを目標としており、2016年4月現在で、およそ13万人の参加を得ています。
また、日本人の個別化医療、個別化予防の実現に向け、ToMMoは、コホート参加者から提供された血液からDNAを抽出し、短鎖型次世代シークエンサーであるHiSeq 2500(Illumina社製)を用いて1,070人の全ゲノム解析を行いました。その結果、約2,120万個の一塩基多様体(SNV)*5を含む日本人の全ゲノムリファレンスパネル(1KJPN)*6の構築をおこないました。この成果は、2014年8月29日に当機構のポータルサイトの一つであるiJGVDから先行して公開*7され、2016年4月1日時点ですでに世界100カ国、1万人以上の方から利用されています。成果は論文化され*8、さらに、2015年12月15日には、SNV約2,120万個すべての情報の公開を行いました。
しかし、短鎖型次世代シークエンサーを用いた解析では、解読できる塩基の長さが数百塩基ずつしかないため、日本人のゲノム配列が持つ数千塩基以上の構造多型を詳細に解明することは困難でした。日本人を対象にしたゲノム医療研究を進めるにあたっては、日本人が独自に有するこれらの構造多型を明らかにしたゲノム配列情報が求められていました。
日本人の基準ゲノム配列の意義
その「お手本」として、国際的組織であるゲノムリファレンスコンソーシアム(Genome Reference Consortium)が管理する国際ヒトゲノム参照配列(以下、国際参照配列)が用いられています。この配列は、日本人の配列情報も一部用いて作成されていますが、欧米人の配列情報を主としており、日本人によく見られる構造多型の情報は含まれていないのではないかと考えられていました。そのことが短鎖型次世代シークエンサーを用いて日本人のゲノム配列を読み取る際の精度に影響するものと考えられていました。
そのため、東北メディカル・メガバンク計画においては、日本人ゲノム配列の高精度化を図るため、参照ゲノムについても日本人独自のもの(日本人の基準ゲノム配列)を作成することといたしました。
日本人の基準ゲノム配列が完成すれば、短鎖型次世代シークエンサーを用いたゲノム解読時の読み取り精度の向上に活用されるとともに、国際参照配列だけを用いても精確に読みとることのできなかった領域の解明に貢献します。それにより、日本人のゲノム情報に基づく疾患解析を行っている研究者が、それぞれの疾患の原因変異をより正確に同定することが可能となると考えています。
このため、日本人の基準ゲノム配列の公開は、我が国のゲノム研究全体を底上げ、加速させるものと期待されます。また、今回の成果は日本人のゲノム配列上の特徴を明らかにする成果であり、今後、日本の医学研究の大きな基盤になると考えられます。
事業の概要
日本人の基準ゲノム配列構築のため、ToMMoは、コホート調査の参加者から提供されたDNAをもとに、長鎖読みとり型次世代シークエンサーPacBioを用いて、全ゲノムを解読しました。
この機器は、平均で1度に1万塩基以上のゲノム配列情報を連続して読むことが可能です。しかし、この機器は読み取りエラーの割合が高いことが知られており、これまで、日本人ゲノムの大規模解析に用いられたことは、ほとんどありませんでした。ToMMoでは、この問題を克服するため、日本人のゲノムDNAから平均1.2万塩基長という長いDNAライブラリーを作成し、一気に読み取ることで大量の配列情報を取得することと、高精度の塩基配列の新規再構成(デノボアセンブル*9)という情報科学的手法を用いることにいたしました。
そのため、長鎖読み取り型次世代シークエンサーを用いて日本人のDNAを繰り返し読み取り、一人あたり合計約3,000億塩基分(ヒトゲノム全体を100回以上繰り返して全て読んだ場合に相当)の配列情報を得ました。また、ToMMoのスーパーコンピュータシステム*10を数ヶ月間にわたって利用して情報解析を行いました。
解析の結果新たに同定された配列の一部を国際ヒトゲノム参照配列上に配した日本人の基準ゲノム配列JRG v1及びデコイ配列decoyJRG v1を構築し、両配列を公開することとしました。
デコイ配列とは、国際ヒトゲノム参照配列には含まれていない領域をまとめたもので、decoyJRG v1は日本人に高頻度でありながら、国際ヒトゲノム参照配列には含まれていない配列ということになります。デコイ配列は主に短鎖型次世代シークエンサーを用いたゲノム解読時の読み取り精度の向上に活用されます。
解読結果の概要
本解読の結果、国際参照配列に対して、日本人が保有しこれまで報告されてこなかった約3,700箇所の新たな挿入配列、約250万塩基の同定に成功しました。
ゲノムリファレンスコンソーシアムは、国際参照配列を管理するとともに、定期的に改定しています。2016年4月現在の最新の国際参照配列は、2013年12月にリリースされたGRCh38です。GRCh38と今回のデノボアセンブルの結果とを網羅的にスーパーコンピュータ上で比較することで、最終的に国際参照配列には収載されていない、約3,700箇所の新たな挿入配列(総塩基として約250万塩基分)の同定に成功しました。
これまで検出できなかった挿入配列を同定可能にした要因は、PacBioが1度に1万塩基以上を連続して読むことが可能な長鎖型次世代シークエンサーであること、さらには、PacBioではDNAをクローニングやPCR増幅することなく1分子ごとに直接観察する手法が用いられていること(これまでの手法では、DNAを複製させる必要がありました)によると考えています。
また、これら挿入配列を短鎖型次世代シークエンサーで構築した1KJPNの日本人集団1,070人および国際1,000人ゲノムプロジェクト*11のうちアフリカ、ヨーロッパ、東アジアなど8集団での存在有無を確認し、一つひとつの挿入配列に対し、他集団にも存在するものとしないものとがあることを見出しました(図1)。
ヒトゲノム中には2万個以上の遺伝子が存在しますが、これらの挿入配列の一部は遺伝子をコードしている領域に含まれており、機能に影響を与えると予想される配列も存在しました。さらに、国際参照配列と比較することで同定できた2,120万個のSNVに加え、今回新規に同定できた約250万塩基の中に5万個以上のSNVが日本人集団に存在することが明らかになりました。
今後の計画
日本人基準ゲノムJRGv1は、近日中に下記のサイトで公開する予定です。なお、今後の研究の進展により、より精細な配列情報が得られた場合は、バージョンアップする予定です。
- 日本人基準ゲノム JRGv1 約30億塩基
- 日本人基準ゲノム用デコイ配列 decoyJRGv1 約250万塩基
用語解説
- *1.コホート調査:
- ある特定の人々の集団を一定期間にわたって追跡し、生活習慣などの環境要因・遺伝的要因などと疾病の関係を解明するための調査。
- *2. 次世代シークエンサー:
- 主に2000年代半ば以降に登場した、DNA配列を高速で読み取る(シークエンス)機器の総称。主に、ランダムに切断されたDNA断片の塩基配列を1塩基ずつ決定する解析過程を、数百万以上ものDNA断片に対して同時並列的に処理することが可能。それまでの解析方法と比較して、精度の高いデータが大量かつ低価格、短時間で得られる。
- *3.国際ヒトゲノム参照配列:
- 国際的な学術組織The Genome Reference Consortiumが継続的に改訂を行っているヒトゲノムの全染色体の塩基配列。同配列は主に欧米の複数のヒトゲノムを読むことで構築されている。事実上、ヒトゲノムのデファクトスタンダードの塩基配列として全世界のヒトゲノム研究に利用されている。平成28年4月現在、最もよく使われている最新の国際ヒトゲノム参照配列はGRCh38である。
- *4.デコイ配列:
- 繰り返し配列などの、短鎖型次世代シークエンサーでの難読領域等を仮想配列として統合した配列。難読配列は、短鎖型次世代シークエンサーによって読みとりはされるが、その読みとり結果を、国際ヒトゲノム参照配列等に照らして並べ(マップし)ようとすると、適合しなかったり、特定箇所に過度に集中するなどして、うまくマップすることができない。そうした領域を、(デコイ=おとりのようにして)人為的に集める仮想配列をつくると、短鎖型次世代シークエンサーの結果の解析に対して有用である。
- *5.一塩基多様体(SNV):
- 個人間でゲノムの一塩基が異なる状態。なお、通常は一定以上の頻度(通常1%)で確認されるSNVを特に一塩基多型(Single Nucleotide Polymorphism)SNPと呼ぶ。
- *6.全ゲノムリファレンスパネル(1KJPN):
- 大規模な人数の全ゲノム解読を行った結果を総合し、DNA配列の多型の頻度などの情報をまとめることで、今後のゲノム研究の参照情報となるよう、東北大学東北メディカル・メガバンク機構が構築を進めている全ゲノムリファレンスパネルのこと。現在、1,070人のコホート参加者の全ゲノム解読により、最終的に信頼度の高い2,120万箇所の一塩基多様体を同定している。
- *7.iJGVD:
- 東北大学東北メディカル・メガバンク機構が公開している、一塩基多様体についてのデータベースのポータルサイトIntegrative Japanese Genome Variation Databaseの略語。アレル頻度5%以上のSNP頻度情報について、一般に公開している。また、誓約事項に同意いただくことで、1KJPNに含まれる、全てのSNVの位置情報、アレル頻度情報およびアレル数情報についてダウンロード可能になっている。
【参考】
- *8.日本人1,070人の全ゲノム解読に関する論文:
- 2015年8月21日、東北メディカル・メガバンク計画のコホート調査事業に参加した宮城県在住の健常な日本人1,070人分 の全ゲノムを解析した成果が英国科学誌「Nature Communications (ネイチャー・コミュニケーションズ)」に掲載された。
【参考】 - *9.デノボアセンブル:
- 断片化されてよみとられた塩基配列の、重複した部分を見つけ出してつなぎ合わせることで、元の染色体の塩基配列での並び順に再構築する情報科学的な手法を指す。今回の場合、概ね1万塩基配列でよみとられた配列から数百万塩基程度の配列(コンティグ)につなぎ合わせることをスーパーコンピュータ上で行った。
- *10.スーパーコンピュータシステム:
- 東北メディカル・メガバンク機構は複合バイオバンクとしてデータバンクおよび解析の機能も併せ持っており、ライフサイエンス分野では日本最大級のスーパーコンピュータシステムの本格運用を有している。
- *11.国際1,000人ゲノムプロジェクト:
- 人類集団の詳細な遺伝的多様性を行うことを目指し、世界各地の約1,000人の全ゲノムシーケンスを行った国際研究計画。当計画は、現在解析人数を2,535人にまで拡張したphase3が完了している。
参考図
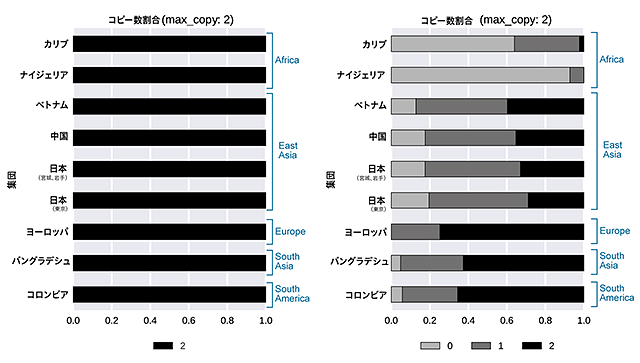 図1 日本人の1,070人でも他集団でも確認されている挿入配列の例(左)、
図1 日本人の1,070人でも他集団でも確認されている挿入配列の例(左)、集団毎に異なる挿入配列の例(右)

図2 日本人基準ゲノムのロゴ
東北メディカル・メガバンク計画について
本計画は、東日本大震災を受け、被災地住民の健康不安の解消に貢献するとともに、個別化予防等の東北発の次世代医療を実現するため、ゲノム情報やオミックス情報を含むコホート研究等を実施し、被災地域の復興を推進する、国の復興事業として行われているものです。2015年度より、国立研究開発法人 日本医療研究開発機構が本計画の研究支援担当機関の役割を果たしています。
被災地に医療関係人材を派遣して地域医療の復興に貢献するとともに、15万人規模の地域住民コホートと三世代コホートを形成し、そこで得られる生体試料、健康情報、診療情報等を収集してバイオバンクを構築します。さらに、ゲノム情報、オミックス情報、診療情報等を解析することで、個別化医療等の次世代医療に結びつく成果を創出することを目指しています。また、得られた生体試料や解析成果を同意の内容等に十分留意し、個人情報保護のための匿名化等の適切な措置を施した上で、外部に提供することや、コホート調査や解析研究を行うための多様な人材の育成も行っています。
本計画の事業の実施は、東北大学東北メディカル・メガバンク機構と岩手医科大学いわて東北メディカル・メガバンク機構とが連携して行っています。
お問い合わせ先
研究に関すること
東北大学東北メディカル・メガバンク機構
シークエンス解析室長
教授 安田 純(やすだ じゅん)
電話番号:022-272-3102
Eメール:jyasuda”AT”megabank.tohoku.ac.jp
東北大学東北メディカル・メガバンク機構
インシリコ解析室長
教授 長崎 正朗(ながさき まさお)
電話番号:022-273-6051
Eメール:nagasaki”AT”megabank.tohoku.ac.jp
報道に関すること
東北大学東北メディカル・メガバンク機構
広報戦略室長
長神 風二 (ながみ ふうじ)
電話番号:022-717-7908
ファックス:022-717-7923
Eメール:f-nagami”AT”med.tohoku.ac.jp
AMED事業に関すること
日本医療研究開発機構(AMED)
バイオバンク事業部 基盤研究課
電話番号:03-6870-2228
Eメール:kiban-kenkyu”AT”amed.go.jp
※Eメールは上記アドレス”AT”の部分を@に変えてください。
掲載日 平成28年4月23日
最終更新日 平成28年4月23日


