プレスリリース ヘミメチルDNAの新機能―NP95の結合により抑制型ヒストン修飾を解除―
プレスリリース
国立研究開発法人日本医療研究開発機構
要旨
理化学研究所(理研)統合生命医科学研究センター免疫器官形成研究グループの古関明彦グループディレクターらの国際共同研究グループは、「NP95[1]」と呼ばれるタンパク質が、転写を抑制するメチル化DNAと転写を活性化する非メチル化DNAの中間状態である「ヘミメチルDNA[2]」に結合することにより、抑制型のH3K9ヒストン修飾[3]を解除し、内在性レトロウイルス(ERV)[4]配列の転写を誘導することを発見しました。今回のへミメチルDNAによる転写制御モデルの発見により、メチル化DNAと非メチル化DNAを中心とした転写制御の従来モデルが大きく見直されました。
ヒトを含むほ乳類ゲノムの約10%は、ERV由来の配列から構成されています。しかし、ERVの異常な転写はゲノムの安定性を揺るがす危険性を伴うため、通常、DNAメチル化やH3K9メチル化などのエピジェネティック修飾[5]によって厳密に抑制されています。これまでの研究により、DNAメチル化機構は、NP95とDNMT1[6]と呼ばれる酵素によって構成されることが知られています。実際にNp95またはDnmt1のどちらの遺伝子をノックアウトしても、DNAメチル化は抑制され、Dnmt1ノックアウトマウスではERVの1種のIAP(intracisternal A particles)の転写が活性化されることが分かっています。一方、長期培養したDnmt1ノックアウト胚性幹(ES)細胞では、IAPの転写が起こらないという不可解な現象が起こります。このことからIAPの転写抑制は、DNAメチル化依存的な機構からH3K9メチル化依存的な機構に切り替わることが示唆されていましたが、詳しいメカニズムは不明でした。
共同研究チームは、Dnmt1およびNp95のノックアウトマウスやES細胞を作製し、IAPの転写抑制におけるDnmt1とNp95の役割を調べました。その結果、Dnmt1ノックアウトマウス細胞の初期段階では、DNAメチル化機構が破綻し、大量のヘミメチルDNAが出現します。しかし、ヘミメチルDNAにNP95が結合することによって、IAPが転写されます。一方、長期培養後のDnmt1ノックアウト細胞では、ヘミメチルDNAがなくなるに伴い、NP95の結合が減少し、下流の「SETDB1-H3K9me3経路」によって、IAPの転写が抑制されることが分かりました。つまり、IAPの転写抑制における分子メカニズムにおいて、DNAメチル化依存的な機構からSETDB1-H3K9me3依存的な機構への移行には、ヘミメチルDNAおよびNP95結合がなくなることが必要であることが分かりました。
本研究により、ヘミメチルDNAおよびNP95を介した新たなエピジェネティック機構を明らかにしました。この機構は、胎盤特異的な遺伝子の転写活性化に寄与している可能性があるため、胎盤機能の異常による流産や不妊の分子メカニズムの解明に役立つと考えられます。
本研究は、日本医療研究開発機構(AMED)の革新的先端研究開発支援事業(AMED-CREST)「エピゲノム研究に基づく診断・治療へ向けた新技術の創出」研究開発領域(研究開発総括:山本 雅之)における研究開発課題「エピジェネティクスによるエンハンサー動態制御メカニズムの解明と細胞機能制御への応用」(研究開発代表者:古関 明彦)の一環で行われました。なお、本研究開発領域は、平成27年4月の日本医療研究開発機構の発足に伴い、国立研究開発法人科学技術振興機構(JST)より移管されたものです。成果は、米国の科学雑誌『Cell Stem Cell』(7月7日号)に掲載されるのに先立ち、オンライン版(4月14日付け:日本時間4月15日)に掲載されました。
※国際共同研究グループ
- 理化学研究所 統合生命医科学研究センター 免疫器官形成研究グループ
- グループディレクター 古関 明彦(こせき はるひこ)
研究員 Jafar Sharif(ジャファル・シャリフ) - ブリティッシュコロンビア大学 Medical Genetics Department
- 教授 Matthew C. Lorincz(マシュー・シー・ローリンクズ)
背景
DNAメチル化やヒストン修飾などのエピジェネティック修飾は、胚発生や細胞の運命決定に深く関わっています。細胞分裂を経て、親細胞から娘細胞にDNAメチル化のパターンが複製されますが、SRAタンパク質[7]の「NP95」はこの過程において中心的な役割を担っています注1、2)。NP95はSRAドメインを介してヘミメチルDNAを認識し、さらにDNAメチル化酵素の「DNMT1」を連れてくることにより、DNAメチル化を忠実に維持できます。
このように、DNAメチル化機構はNP95およびDNMT1によって構成されているため、Np95またはDnmt1のどちらの遺伝子を欠損しても、DNAメチル化が低下します。実際に、Dnmt1を欠損した発生中期のマウス(胎児)では、IAPという「内在性レトロウイルス(ERV)」が劇的に転写活性化します注3)。しかし不可解なことに、長期間培養したDnmt1ノックアウトES細胞では、IAPの転写がほとんど起こりません注4)。長期培養後のDnmt1ノックアウトES細胞では、“H3K9メチル化による抑制型クロマチン修飾が、DNAメチル化の代わりにIAPの転写を阻止している”というモデルが、その後に提唱されています注5)。
IAPの転写を抑制するエピジェネティック経路が、どのようにしてDNAメチル化依存的なものからH3K9me3依存的なものに切り替わるのか、詳しいメカニズムは分かっていません。NP95はDNAメチル化およびH3K9me3注6)の両方ともを認識するため、IAP転写に重要な役割を持つ可能性があります。
そこで、国際共同研究グループはNP95に着目し、IAPの転写抑制におけるDNAメチル化およびH3K9me3の相互作用の解析に挑みました。
- 注1)
- 2007年11月30日プレスリリース「エピジェネティックな遺伝情報発現の制御機構を発見」
- 注2)
- Sharif J, Muto M, Takebayashi S, Suetake I, Iwamatsu A, Endo TA, Shinga J, Mizutani-Koseki Y, Toyoda T, Okamura K, Tajima S, Mitsuya K, Okano M & Koseki H. The SRA protein Np95 mediates epigenetic inheritance by recruiting Dnmt1 to methylated DNA. Nature. 450, 908-12.(2007)
- 注3)
- Walsh C P, Chaillet J R & Bestor T H. Transcription of IAP endogenous retroviruses is constrained by cytosine methylation. Nat Genet. 20, 116-7.(1998)
- 注4)
- Hutnick L K, Huang X, Loo T C, Ma Z & Fan G. Repression of retrotransposal elements in mouse embryonic stem cells is primarily mediated by a DNA methylation-independent mechanism. J Biol Chem. 285, 21082-91.(2010)
- 注5)
- Matsui T, Leung D, Miyashita H, Maksakova I A, Miyachi H, Kimura H, Tachibana M, Lorincz M C & Shinkai Y. Proviral silencing in embryonic stem cells requires the histone methyltransferase ESET. Nature 464, 927-31.(2010)
- 注6)
- Arita K, Isogai S, Oda T, Unoki M, Sugita K, Sekiyama N, Kuwata K, Hamamoto R, Tochio H, Sato M, Ariyoshi M & Shirakawa M. Recognition of modification status on a histone H3 tail by linked histone reader modules of the epigenetic regulator UHRF1. Proc Natl Acad Sci U S A. 109, 12950-5.(2012)
研究手法と成果
国際共同研究グループは、Dnmt1およびNp95のノックアウトマウスやES細胞を作製し、IAPの転写抑制におけるDNAメチル化因子の役割を解析しました。Dnmt1ノックアウトマウスおよびES細胞では、既に報告されているように、IAPが劇的に転写されていました。しかし驚くべきことに、Dnmt1ノックアウトと同様に、DNAメチル化が低下するNp95ノックアウトでは、IAPの転写がほとんど上昇しませんでした(図1)。さらに、Dnmt1およびNp95の両因子ともをノックアウトしたマウスおよびES細胞でも、IAPの転写活性化がみられませんでした。これらの結果から、IAPの転写活性化はDNA脱メチル化に依存しないと考えられ、さらに、NP95がIAPの転写に何らかの役割を持っていることが示唆されます。
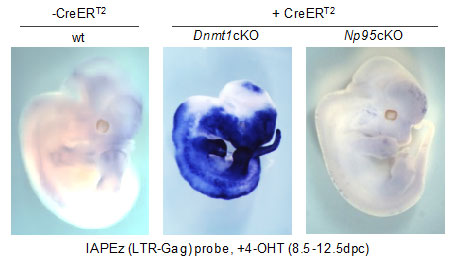 図1 Dnmt1、およびNp95ノックアウトマウス胎児におけるIAP転写
図1 Dnmt1、およびNp95ノックアウトマウス胎児におけるIAP転写
左:野生型のマウス胎児
中:Dnmt1ノックアウトマウス胎児。劇的なIAP転写(青色で標識)が起きている。
右:Np95ノックアウトマウス胎児。DNAメチル化が同様に低下するにも関わらず、IAP転写が起きない。
Dnmt1ノックアウトマウス細胞ではDNAメチル化が破綻するため、遅延性のヘミメチルDNA[8]が出現します。国際共同研究グループは、このような遅延性のヘミメチルDNAにNP95が“通常よりも長く結合する”ことにより、下流のH3K9me3の蓄積が阻害されることを発見しています。ヒストンメチル化酵素のSETDB1は、IAPを含むERV領域へのH3K9me3蓄積を媒介しますが、Np95ノックアウトES細胞では、この「SETDB1-H3K9me3経路」によってIAPの転写が抑制されます。実際に、Np95とSetdb1の両遺伝子を同時にノックアウトすると、予想通り、IAPが劇的に転写活性化したことから、NP95とSETDB1との間に阻害的な関係が存在することが考えられます(図2)。
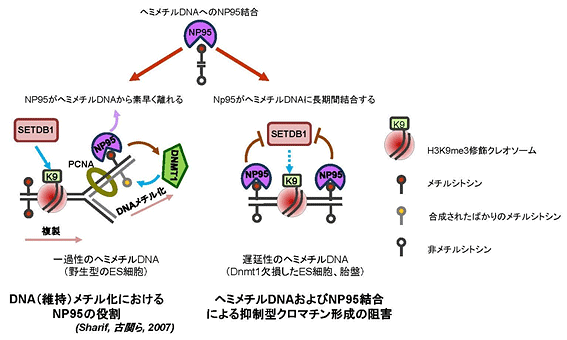 図2 一過性と遅延性のヘミメチルDNAへのNP95の結合及びその生物学的な意義
図2 一過性と遅延性のヘミメチルDNAへのNP95の結合及びその生物学的な意義
一過性のヘミメチルDNAへのNP95の結合は、DNMT1を介してフルメチル化(2鎖DNAの両方の鎖がメチル化された)状態を誘導する(左)。一方、遅延性のヘミメチルDNAにNP95が結合すると、SETDB1-H3K9me3による抑制型のクロマチン修飾が阻害される(右)。
これらの結果をまとめると、Dnmt1をノックアウトした初期段階の細胞では、DNAメチル化機構の破綻により、大量のヘミメチルDNAが出現しますが、ヘミメチルDNAにNP95が結合することによってIAPが転写されます。しかし、長期培養後のDnmt1ノックアウトマウス細胞では、ヘミメチルDNAがなくなるのに伴い、NP95の結合が減少するため、SETDB1-H3K9me3機構によってIAPの転写が抑制されます。つまり、IAPの転写抑制における分子メカニズムにおいて、DNAメチル化依存的な機構からSETDB1-H3K9me3依存的な機構へと移行するためには、ヘミメチルDNAおよびNP95結合がなくなることが必要であると考えられます。
マウスの体細胞や胎児では、IAPを含むERV配列がDNAメチル化やH3K9me3化などの複数のエピジェネティック機構によって転写抑制されていることが、過去の研究から明らかになっています。ERVの転写はゲノムの安定性に悪影響を及ぼすため、DNAメチル化やH3K9me3などの修飾は細胞防御システムの一環として、ERVの転写を抑制すると考えられています。国際共同研究グループの研究から、胎盤などの胚体外組織ではIAPが高いレベルで転写されていることが発見されています。これは、胎盤はERV転写を許容していることを意味し、さらに、胎盤の発生や正常な機能にはERV転写の必要性があることを示唆しています。興味深いことに、Np95をノックアウトしたマウスの胎盤ではIAPの転写が減少します。つまり胚体外組織では、ヘミメチルDNAへのNP95の結合がIAPなどの配列からの転写活性化を制御していると思われます。
今後の期待
本研究は、ヘミメチルDNAおよびNP95を介した新たなエピジェネティック機構を明らかにしました。この機構は、胎盤特異的な遺伝子の転写活性化に寄与している可能性があるため、胎盤機能の異常による流産や不妊の分子メカニズムの解明に役立つと思われます。
同様のエピジェネティック機構は、5-Aza-dCなどの低分子化合物の持つ、がん抑制効果にも寄与している可能性があります。5-Aza-dCは、DNAの維持メチル化を阻害するため、遅延性のヘミメチルDNAの蓄積を誘導します。これらのヘミメチルDNAにNP95が結合すると標的ゲノム領域からの転写が活性化されると予想されます。
さらに、NP95とSETDB1との阻害的な関係は、初期発生において重要な機能を持っていると思われます。ほ乳類の受精卵や2細胞胚などでは、NP95のほとんどが細胞質に輸送されており、核内では「Np95欠損」に類似した状況に置かれています。この時期には、ゲノムが低DNAメチル化状態になっていますが、H3K9me3機構がDNAメチル化の代わりにERVを抑制していることが、近年の研究から明らかになっています。NP95が核外に輸送されていることで、H3K9me3による円滑なIAP抑制が起きている可能性が示唆され、ほ乳類の初期発生におけるエピゲノム制御を理解するための重要な知見となります。
論文情報
- タイトル
- Activation of Endogenous Retroviruses in Dnmt1–/– ESCs Involves Disruption of SETDB1-Mediated Repression by NP95 Binding to Hemimethylated DNA
- 著者名
- Jafar Sharif, Takaho A. Endo, Manabu Nakayama, Mohammad M. Karimi, Midori Shimada, Kayoko Katsuyama, Preeti Goyal, Julie Brind'Amour, Ming-An Sun, Zhixiong Sun, Tomoyuki Ishikura, Yoko Mizutani-Koseki, Osamu Ohara, Yoichi Shinkai, Makoto Nakanishi, Hehuang Xie, Matthew C. Lorincz and Haruhiko Koseki
- 雑誌
- Cell Stem Cell
- DOI
- http://dx.doi.org/10.1016/j.stem.2016.03.013
補足説明
- [1]NP95
- NP95は、Nuclear Protein 95 kDaの略。メチル化した遺伝子プロモーター領域(遺伝子を発現させる機能を持つ塩基配列)に結合するほ乳類のタンパク質。DNAの修復や細胞周期の進行に重要といわれている。UHRF1とも呼ばれる。
- [2]ヘミメチルDNA
- ヘミメチルDNA は、2本鎖DNAの片方の鎖のみがメチル化された状態のDNA。複製後もこの状態を保持する機構が働く。一方、フルメチルDNAは2本鎖DNAの両方の鎖がメチル化された状態のDNAのこと。
- [3]H3K9ヒストン修飾
- ヒストンはDNAとの親和性の高い塩基性タンパク質。主に4種のヒストン(2A、H2B、H3、H4)がそれぞれ2分子ずつ集まって8量体を形成し、この周りにDNAが巻き付いてクロマチン構造の最小単位「ヌクレオソーム」を形成する。ヌクレオソームからは各ヒストンのアミノ酸末端領域が突出しており、アセチル化、メチル化などの修飾を受けて、クロマチン構造が弛緩、凝縮し、遺伝子発現が制御される。これをヒストン修飾と呼ぶ。H3K9ヒストン修飾は、ヒストンH3の9番目のリジン残基(K)を修飾することを指す。
- [4]内在性レトロウイルス(ERV)
- 生物は進化の過程において、ウイルスなどの外来性のDNA源によって繰り返し侵略されてきた。ゲノムがRNAで構成されているレトロウイルスの場合は、宿主の細胞環境を利用して自らのタンパク質を合成するには、逆転写によりRNAからcDNA(相補的DNA)を合成する必要がある。逆転写されたcDNAが宿主の生殖細胞ゲノムに挿入されてしまうと遺伝情報の一部として次世代に受け継がれることになる。このように、宿主ゲノムに寄生するレトロウイルス性DNA配列は「内在性レトロウイルス(ERV)」と呼ばれる。ヒトを含むほ乳類の場合は、ゲノム全体の約10%がERV配列から構成されている。ほとんどのERV配列には、点変異や欠損が起きているため、ウイルス性のタンパク質を合成する機能が失われている。さらに、DNAメチル化やH3K9me3などの抑制型エピジェネティック修飾によってERV配列からの転写そのものが厳密に抑えられている。ERVはEndogenous Retrovirusesの略。
- [5]エピジェネティック修飾
- DNAの塩基配列の変化を伴わず、遺伝子の発現を活性化したり不活性化したりする後付けの修飾のこと。主たる現象として、DNAのメチル化修飾、ヒストンのアセチル化やメチル化、リン酸化が知られる。正常な発生や分化に関わる重要な機構で、その破綻は、さまざまな発生・分化異常やそれに伴う疾病を引き起こし、最近では、がん治療や再生医療において重要なテーマになりつつある。
- [6]DNMT1
- ゲノムにおけるDNAメチル化パターンの維持に必須なDNAメチル基転移酵素。DNA複製に伴って生じる肩側DNA鎖だけがメチル化された部分において、メチル化されていないほうのDNA鎖に新たなメチル基を導入する。
- [7]SRAタンパク質
- SRAドメインは、非対称性DNAメチル化(asymmetric DNA methylation)を認識するアミノ酸モチーフである。SRAドメインを持つタンパク質は、SRAタンパク質という。SRAドメインは最初に植物で発見されたが、その後、脊椎動物でも保存されていることが明らかになっている。哺乳類ではこれまで、Np95/Uhrf1とNp97/Uhrf2との2つのSRAタンパク質が発見されている。SRAはSET and RING-finger Associated Domainの略。
- [8]遅延性のヘミメチルDNA
- ヘミメチルDNAは、フルメチルDNA(fully methylated DNA)と非メチルDNA(unmethylated DNA)の中間体として考えられている。通常、DNAの維持メチル化の際に、複製フォークに一過性のヘミメチルDNAが出現するが、NP95およびDNMT1の働きによってすぐにフルメチルDNAに変換される。NP95がSRAドメインを介して一過性のヘミメチルDNAを認識し、DNMT1をリクルートする。しかし、NP95がヘミメチルDNAに結合したままでいると、(ヘミメチルDNAを含む)DNA二重らせんの内部にDNMT1が入り込むことができない。そのため、NP95はDNMT1をリクルートしたらすぐにヘミメチルDNAから離れる。一方、Dnmt1ノックアウトES細胞では、DNMT1が欠損しているため、複製に依存したDNAの維持メチル化機構が破たんし、数回の細胞分裂を経ても持続するヘミメチルDNAが出現する。このように、長期間において存在するヘミメチルDNAは「遅延性の(protracted)ヘミメチルDNA」と呼ばれる。DNMT1cKO ES細胞では、NP95がこれらの遅延性のヘミメチルDNAに結合したままでいるため、結果的に、下流のSETDB1を介した抑制型クロマチン修飾が阻害される。
お問い合わせ先
研究内容について(発表者)
理化学研究所 統合生命医科学研究センター
免疫器官形成グループ
グループディレクター 古関 明彦(こせき はるひこ)
TEL:045-503-9690
FAX:045-503-9688
E-mail:haruhiko.koseki“AT”riken.jp
広報について
理化学研究所 広報室 報道担当
TEL:048-467-9272 FAX:048-462-4715
E-mail:ex-press“AT”riken.jp
日本医療研究開発機構の事業について
日本医療研究開発機構(AMED)
戦略推進部 研究企画課
TEL:03-6870-2224 FAX:03-6870-2243
E-mail:kenkyuk-ask“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
掲載日 平成28年4月27日
最終更新日 平成28年4月27日


