プレスリリース 独自の数理モデルから、生きているDNAの構造情報の取得に成功
プレスリリース
国立大学法人広島大学
国立遺伝学研究所
国立研究開発法人日本医療研究開発機構
本研究成果のポイント
- 細胞の核内で折りたたまれたひも状のDNAを表現する数理モデルを考案し、その動きと構造(ひもの形)の関係を表す理論式を導き出した。
- ヒトの生細胞でDNAにつけた蛍光分子の動きを精密に計測し、上の理論式を用いることで、生きている状態でのDNAの構造情報を得ることに成功した。
- 本研究での数理モデルに基づく解析を展開することで、クロマチンの動きと対応する遺伝子発現調節機構の関係の解明につながり、数理科学的アプローチによる遺伝子発現の予測や創薬への応用に役立つと期待される。
概要
広島大学大学院理学研究科・クロマチン動態数理研究拠点の新海創也特任助教らの研究グループは、ヒトの生細胞の核内クロマチン(注1)の動きを表現する数理モデルを考案しました。実際に、その動きを精密に観測したところ、遅くて異常な動きはクロマチンドメイン(注2)の構造に拘束された動きであることを突き止めました。また、生きている状態のクロマチンドメインの大きさとフラクタル次元(注3)といった構造情報を得ることに成功しました。
本研究成果は、「PLOS Computational Biology」オンライン版に10月20日(米国東部標準時間)に掲載されます。
掲載雑誌:PLOS Computational Biology
DOI番号:10.1371/journal.pcbi.1005136
論文題目:Dynamic Nucleosome Movement Provides Structural Information of Topological Chromatin Domains in Living Human Cells
著者:Soya Shinkai*, Tadasu Nozaki, Kazuhiro Maeshima and Yuichi Togashi *Corresponding author(責任著者)
背景
ヒトのDNAは全長2メートルもあり、それがわずか10マイクロメートル(1メートルの10万分の1の大きさ)の細胞核の中に階層的に格納されています。近年その格納のされ方に関して、ゲノムの空間的平均構造を明らかにした染色体構造捕獲法(注4)の技術のおかげで、遺伝子発現調節の基本構造単位となる数Mbサイズ(数百万のDNAの文字列)の領域(ドメイン)が注目を集めています。しかし、このドメインに関する知見は、細胞核のDNAを固定化して、数億個の細胞から回収されたDNA断片のデータから得られたものであり、生きている状態でのドメインの構造に関する情報はこれまで全く知られていませんでした。
一方、本研究グループの情報・システム研究機構国立遺伝学研究所の前島一博教授、野崎慎研究員らは、これまでに、生きている細胞の核内で、DNAを格納する微細構造であるヌクレオソーム(注5)の動きを精密に観測することに成功してきました。そして、その動きはブラウン運動のように、たくさんの分子の熱ゆらぎからの影響に支配されていることを見出してきました。しかし、その動きはブラウン運動よりもずっと遅くて異常な動きであり、それを説明するための理論はありませんでした。
研究成果の内容
今後の展開
※用語解説
- (注1)クロマチン
- 核内に存在するDNAとタンパク質からなる複合体。
- (注2)クロマチンドメイン
- 遺伝子発現調節の基本構造単位となる数Mbサイズ(数百万のDNAの文字列)のクロマチン領域が核内で形成するかたまり(クラスター)。
- (注3)フラクタル次元
- 海岸線や木の葉、雪の結晶のような複雑で自己相似的な形状は、しばしばフラクタル次元を用いて表現することができる。ここでは、数珠状のヌクレオソームファイバーからなるクロマチンドメインの疎密状態を特徴づける指標として用いている。
- (注4)染色体構造捕獲法
- 核内で近接しているゲノム領域を特定する方法のことで、通称3C(Chromosome Conformation Capture)法と呼ばれる。この派生として4C、5C、Hi-Cといったものがある。
- (注5)ヌクレオソーム
- 真核生物の核内クロマチンの基本構成単位であり、タンパク質(ヒストン)でできたコアにDNAが約2周分巻きついて構成されている。
研究支援
参考資料
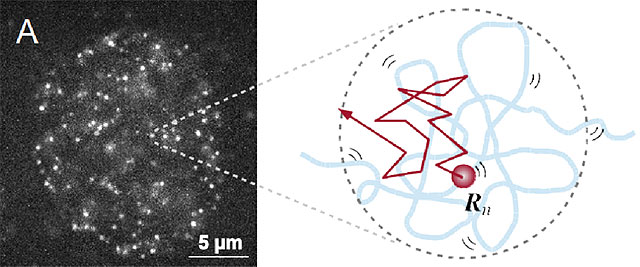
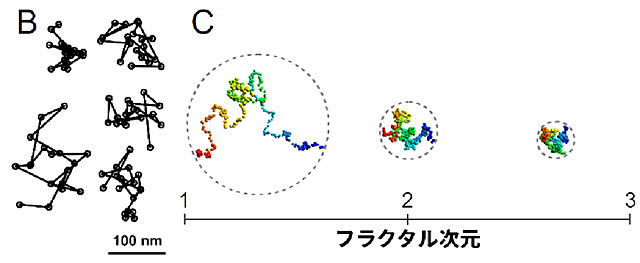
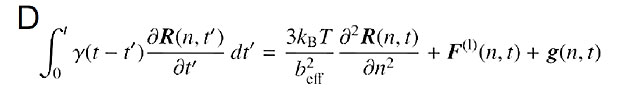
(A)核内のDNAにつけた蛍光分子が自発的にランダムに光っている様子をとらえた顕微鏡画像(左)。最近の染色体構造捕獲法の知見を踏まえると、画像の輝点はクロマチンドメイン内の一つのヌクレオソームに対応すると考えられる(右)。0.05秒毎に画像を取得することで、その輝点が動いていることがわかった。しかしながら、クロマチンドメインの全貌はわからない。(B)約1秒間での典型的なヌクレオソームの軌道が5つ描かれている。100ナノメートルの領域をランダムに動く。(C)クロマチンドメインの疎密の度合いは1~3の間の値をもつフラクタル次元を使って特徴づけることができる。(D)核内ヌクレオソームの動きを説明する数式。
お問い合わせ
本研究成果に関するお問い合わせ先
大学院理学研究科・クロマチン動態数理研究拠点 特任助教 新海 創也
Tel:082-424-5529 FAX:082-424-5529
E-mail:soya“AT”hiroshima-u.ac.jp
AMED事業に関するお問い合わせ先
国立研究開発法人日本医療研究開発機構(AMED)戦略推進部医薬品研究課
〒100-0004 東京都千代田区大手町1-7-1
Tel:03-6870-2219
E-mail:20-DDLSG-16“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
掲載日 平成28年10月21日
最終更新日 平成28年10月21日


