プレスリリース 小児T細胞性急性リンパ性白血病における 極めて高い悪性度に関連する融合遺伝子を発見 ―PU.1/SPI1融合遺伝子―
プレスリリース
国立大学法人京都大学
国立研究開発法人日本医療研究開発機構
発表者
関 正史 (東京大学医学部附属病院 小児科 助教)
木村 俊介 (東京大学医学部附属病院 小児科 登録研究員/広島大学大学院医歯薬保健学研究科小児科学大学院生)
発表のポイント
- 小児T細胞性急性リンパ性白血病(T-ALL)において、極めて高い悪性度に関連する遺伝子異常として、PU.1/SPI1融合遺伝子を世界で初めて複数例に同定しました。
- 大規模シークエンスによるがんゲノム(遺伝情報)の網羅的な解読を行い、小児T-ALLの遺伝子異常、ゲノムの構造変化、遺伝子発現の全体像を明らかにしました。
- 新規SPI1融合遺伝子を有するT-ALLは、遺伝学的特性と臨床的特性がその他のT-ALLとは大きく異なることが明らかとなり、正確な分子診断による治療の適正化に役立つものと期待されます。
発表概要
白血病(注1)は血液中の細胞のうち、白血球になるもとの細胞から発生する悪性腫瘍です。小児期の悪性腫瘍の中では最も高頻度に発生し、T-ALLは小児白血病の約15%を占めています。薬物療法を中心とした集学的治療の強化により全体として約70%の治癒が期待できますが、小児では特に成長障害、臓器機能障害、不妊など、治療後に発生する障害(晩期障害)が大きな課題となっています。また、治療抵抗例や再発した場合の治癒は極めて難しいのが現状です。従って、分子病態に立脚した治療の最適化は、小児T-ALL患者さんの治癒率改善と重篤な副作用や晩期障害の回避に重要といえます。
東京大学医学部附属病院小児科の滝田順子准教授、関正史助教、木村俊介研究員らは京都大学大学院医学研究科腫瘍生物学講座の小川誠司教授らと共同で、次世代シーケンサー(注2)技術を用いて小児T-ALL 123例のゲノム(注3)上にみられる遺伝子異常や融合遺伝子(注4)を含む構造変化、遺伝子発現の異常の全体像を解明しました。その結果、極めて高い悪性度に関連するSPI1(注5)融合遺伝子を約4%の例に同定しました。SPI1融合遺伝子は、T細胞の分化の停止と細胞増殖をもたらし、それが白血病化を引き起こす可能性を示しました。また遺伝子発現パターンと分子学的特徴から小児T-ALLは5群に分類されることを見出し、それぞれの群を特徴づける遺伝子発現や遺伝子異常と臨床的特性を明らかにしました。SPI1融合遺伝子を有する群は、他とは異なる特徴的な一群であることを示し、新たなT-ALLのサブグループであることを示しました。この成果は、T-ALLの予後予測、精度の高い分子診断法の開発に貢献し、治療の最適化の実現に役立つものと期待されます。
本研究は、文部科学省「次世代がん医療創生研究事業(P-CREATE)」の一環として行われたものであり、その成果は2017年7月4日午前0時(英国時間7月3日午後4時)にNature Geneticsのオンライン版で公開されます。
発表内容
研究の背景・先行研究
白血病は小児期の悪性腫瘍の中では最も高頻度に発生する血液を作る骨髄やリンパ節などの造血組織に発生する悪性腫瘍です。T-ALLは小児白血病の約15%を占めています。薬物療法を中心とした集学的治療の強化により全体として約70%の治癒が期待できますが、非寛解や再発した場合は治癒が極めて困難となります。また救命された例では、化学療法の副作用による不妊や成長障害、臓器機能障害などが重大な問題であり、現在の治療は、将来のある小児の患者さんにとって十分であるとは言えません。日本では地区ごとに東京小児がん研究グループ(TCCSG)、小児白血病研究会(JACLS)などの治療研究グループを中心とした臨床試験を行っていましたが、現在は日本小児がん研究グループ(JCCG)というグループに統一されて全国規模の臨床試験を行っています。総じて、予後の悪い群に対する新規治療法の開発や、精度の高い予後予測に基づいた治療の軽減は重要な課題です。そのため、T-ALLの発症や進展にはどのような分子機構が関わっているかを解明する必要があります。
T-ALLは、免疫をつかさどるT細胞の分化成熟の過程の異常により発症すると考えられています。T細胞の成熟は胸腺と呼ばれる臓器で行われます。もっとも幼弱なT-ALLは初期前駆T細胞性ALL(Early T-cell Precursor ; ETP-ALL)と呼ばれ、次世代シーケンサーによる網羅的ゲノム解析により、転写因子系の変異を多く有することが報告されました。また、より成熟したT-ALLでは、TAL1やTLX遺伝子などの異常な発現がみられることも報告されています。しかし、これらは少数例での報告であり、大規模な研究による分子病態の全貌の解明はされていませんでした。さらに、T-ALLでは予後不良を示す分子異常やバイオマーカーも見つかっていないのが現状です。
研究内容
そこで、研究グループはゲノム異常と遺伝子発現の異常を同定する網羅的ゲノム解析を行い、予後の極めて不良な例に重複するSPI1融合遺伝子を世界で初めて報告しました。さらに臨床的な情報も加えた解析によりT -ALLにおけるゲノム異常の全体像を解明しました。
具体的には、T-ALL計121例から腫瘍RNAを採取し、全トランスクリプトーム解析(注6)により融合遺伝子の検索を行いました。過去に報告された融合遺伝子に加えて、新しくSPI1融合遺伝子を5例に検出しました(図1)。さらに60例のT-ALLを追加解析し、計7例(3.9%)に新規融合遺伝子を検出しました。続いてSPI1融合遺伝子を強制的に発現させた機能解析を行い、SPI1融合遺伝子が野生型のSPI1と同様の機能を持つこと、強い細胞増殖能を有すること、T細胞の成熟を途中でとめてしまうことを培養細胞とマウスを用いた実験で証明しました(図2)。また、クラスタリング(注7)という手法を用いて、遺伝子発現のプロファイリングを行い、123例のT-ALLを発現プロファイルの異なる5群に分類しました。既知の4群(①ETP群、②TAL1高発現type A; TAL1-RA、③TAL1高発現type B; TAL1-RB、④TLX関連群)に加えて、他のどの群とも異なる発現パターン(FLT3やKIT高発現)を呈するSPI1融合遺伝子の症例のみで形成される群を検出しました。一方、ALLで変異の多くみられる158遺伝子とその近傍領域に対する変異解析(ターゲットキャプチャー解析(注8))を行い、SPI1融合遺伝子群でRAS経路の異常が多くみられることを明らかにしました(図3)。また、予後との関連についても検討し、SPI1融合遺伝子群では発病後3年以内に7例中6例(85%)が早期再発により死亡しており、他の群のT-ALLと比較して著しく予後が悪いことを見出しました(図4)。これまで予後不良を示すバイオマーカーが存在しなかったT-ALLにおいて、予後不良と関連するSPI1融合遺伝子を報告したことは、今後、この融合遺伝子が予後不良バイオマーカーとなりうる点において、臨床的なインパクトも大きいと言えます。さらにその分子学的特性を明らかにしたことで、SPI1融合遺伝子を有する症例に対してRAS経路を標的とした治療やFLT3やKIT阻害薬を用いた治療戦略の可能性が示され、今後の治療を行う上で非常に有用と考えられます。
社会的意義・今後の予定
本研究によって日本人における小児T-ALLのゲノムの全体像が明らかになっただけでなく、極めて高い悪性度と関連するSPI1融合遺伝子の特性も明らかとなり、T-ALLの分子病態の理解と新たな治療戦略の可能性に大きな進展をもたらしました。T-ALLは既知の4群に、今回新たに検出した新規SPI1融合遺伝子で構成される群を加えた5群に分類されることが判明しました。これは新規SPI1融合遺伝子がその他のT-ALLとは明らかに異なる特性を持つサブグループであることを示しています。さらにこの群が極めて予後不良であることや、分子標的薬を用いた治療戦略の可能性があることも考慮すると、新規分子診断法の提唱という観点から非常に重要な発見であるといえます。この新たな分子診断は、患者さんの個々の治療を考える上で重要であり、個別化医療の実現に多大な貢献をなすものと考えられます。精度の高い新規分子診断は今後のT-ALLの治療の最適化に貢献し、治癒率を改善するのみならずQOLの向上にもつながるものと期待されます。今後はエピゲノムの解析を加えた統合的解析を行い、さらなる治療標的やバイオマーカーの同定を目指します。
本研究は、TCCSG、JACLSを中心に多施設より検体提供をうけ、東京大学医学部附属病院小児科の滝田順子准教授、京都大学大学院医学研究科腫瘍生物学講座の小川誠司教授、群馬県立小児医療センターの林泰秀前病院長、東京大学医科学研究所附属ヒトゲノム解析センターの宮野悟教授らの共同研究チームによって行われました。
発表雑誌
- 雑誌名:
- Nature Genetics
- 論文タイトル:
- Recurrent PU.1/ SPI1 fusions in high-risk pediatric T cell acute lymphoblastic leukemia
- 著者:
- Masafumi Seki, , Shunsuke Kimura, Tomoya Isobe, Kenichi Yoshida, Hiroo Ueno, Yaeko Nakajima-Takagi, Changshan Wang, Lin Lin, Ayana Kon, Hiromichi Suzuki, Yusuke Shiozawa, Keisuke Kataoka, Yoichi Fujii, Yuichi Shiraishi, Kenichi Chiba, Hiroko Tanaka, Teppei Shimamura, Kyoko Masuda, Hiroshi Kawamoto, Kentaro Ohki, Motohiro Kato, Yuki Arakawa, Katsuyoshi Koh, Ryoji Hanada, Hiroshi Moritake, Masaharu Akiyama, Ryoji Kobayashi, Takao Deguchi, Yoshiko Hashii, Toshihiko Imamura, Atsushi Sato, Nobutaka Kiyokawa, Akira Oka, Yasuhide Hayashi, Masatoshi Takagi, Atsushi Manabe, Akira Ohara, Keizo Horibe, Masashi Sanada, Atsushi Iwama, Hiroyuki Mano, Satoru Miyano, Seishi Ogawa, & Junko Takita
- DOI番号:
- 10.1038/ng.3900
用語解説
- 注1:白血病
- 血液をつくる骨髄において、血液細胞を作る(造血)過程で生じた血液のがん。白血病細胞が異常に増加して骨髄を占拠することで正常の造血が阻害されるため、感染症、貧血、出血症状などの症状が出現する。がん化する血液細胞の種類により大きく「骨髄性」と「リンパ性 (ALL)」に、病気の進行パターンや症状により「急性」と「慢性」に分類される。リンパ性白血病はリンパ球の種類によりB細胞とT細胞に分かれる。本論文ではT細胞性急性リンパ性白血病 (T-ALL)について解析を行った。
- 注2:次世代シーケンサー
- 大量同時並列シーケンサーとも呼ばれ、従来の手法と比較し極めて高速にDNAの塩基配列を読むことが可能となり、スーパーコンピューターを用いれば、ヒト全ゲノムの解析もわずか1週間以内で解読が可能となった。
- 注3:ゲノム
- ある生物のもつすべての遺伝情報、あるいはこれを保持するDNAの全塩基配列のこと。タンパク質のアミノ酸配列をコードするコーディング(エクソン)領域とそれ以外のノンコーディング領域に大別される。
- 注4:融合遺伝子
- 染色体・ゲノムの組換えの結果、複数の遺伝子が連結されて生じる新たな遺伝子で、融合タンパク質をコードするもの。融合遺伝子は細胞機能に影響を与えないものも多いが、異常な遺伝子発現をもたらした場合、がん化の原因となりうる。
- 注5:SPI1遺伝子
- SPI1遺伝子はPU.1タンパクをコードしている。PU.1は転写因子のひとつである。正常の血液細胞を作る過程においてPU.1が発現すると、T細胞への分化は抑制され、B細胞や骨髄性細胞へと分化がすすむ。
- 注6:全トランスクリプトーム解析
- 遺伝子転写産物(mRNA)すべてを、次世代シーケンサーを用いて網羅的に解析する解析手法。融合遺伝子の検出だけでなく、生体細胞内における遺伝子発現の状況を網羅的に評価することが可能である。
- 注7:クラスタリング
- 複数サンプルを大きくいくつの群に分かれるのか推定することができる手法。複数のサンプルのうち、一番傾向が似ている2つのサンプルを横に並べていく。さらに2つのサンプルの結果を平均化し、同様に傾向が似ているサンプルを横に並べていくと、トーナメント表のような樹形図を書くことができる。
- 注8:ターゲットキャプチャー解析
- 全ゲノム領域のうち、あらかじめ指定した領域の配列のみを次世代シーケンサーを用いて網羅的に解析する手法。全ゲノム領域を解析する全ゲノム解析や全エクソン領域を解析する全エクソン解析と比較して、解析する領域を必要な領域にしぼりこむことによって低コストで大量の検体を解析することが可能となる。近年行われ始めたクリニカルシークエンスも同様の手法が用いられている。
添付資料
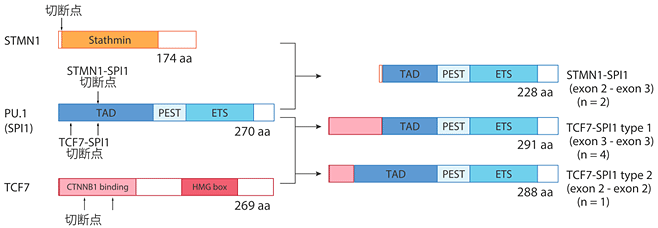
図1 新規SPI1融合遺伝子のシェーマ
左側に各遺伝子がコードするタンパクの構造を、右側に融合遺伝子の構造を示す。内部の区画はタンパク内のドメインを意味している。矢印は融合遺伝子形成時における切断点を示しており、STMN1/TCF7の各切断点とPU.1(SPI1)の切断点が融合して新規SPI1融合遺伝子となる。本研究ではSTMN1-SPI1、TCF7-SPI1(2種類)の計3種類のSPI1融合遺伝子を検出した。
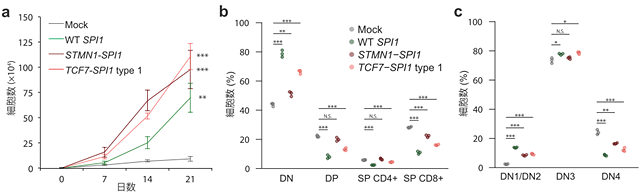
図2 新規SPI1融合遺伝子による細胞増殖と分化への影響
(a)細胞内で野生型SPI1とSPI1融合遺伝子を強制発現させたところ、Mock(コントロール)と比較してSPI1融合遺伝子の発現により著しい細胞増殖能を示した。(b, c)胸腺を模したTSt4-DLL1系を用いて野生型SPI1とSPI1融合遺伝子を強制発現したマウスの幼弱胸腺細胞(DN1/2)の分化能を調べた。正常胸腺細胞は、DN1、DN2、DN3、DN4、DPという順に成熟し、SP CD4陽性細胞とSP CD8細胞に分化しする。Mockとは異なりT細胞の分化が進まず(DP、SP CD4/8+が少ない)、DNで分化が停止している(幼弱胸腺細胞が相対的に多い)ことがわかった。さらにDNの中でもより幼弱なDN1/2で分化が停止している(DN1/2が相対的に多く、DN4が相対的に少ない)ことがわかった。
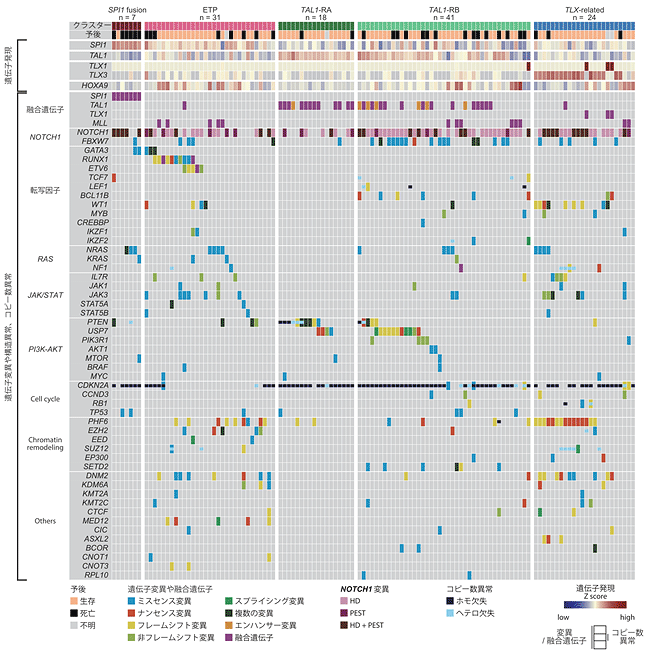
図3 小児T-ALLのクラスタリングと変異やコピー数異常の全体像
T-ALLは発現によるクラスタリングにより5群に分類された。過去に報告されていたETP、TAL1-RA、TAL1-RB、TLXの4群に加えて、今回新たに報告した新規SPI1融合遺伝子のみで形成される群が認められた。この群はCDKN2Aの欠失に加えて、NOTCH1やRAS系の変異が多くみられた一方で、ETP群では転写因子系の変異が、TAL1群ではPTENやUSP7変異が、TLX群ではPHF6変異が多く検出された。
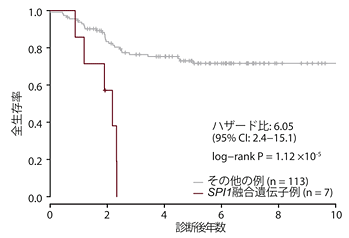
図4 新規SPI1融合遺伝子例とその他のT-ALL症例の生存期間の関係
生存期間について解析するとSPI1融合遺伝子例において明らかに死亡例が多く、全8例中7例が診断後3年以内に死亡していることがわかった。
問い合わせ先
研究に関するお問い合わせ
東京大学医学部附属病院 小児科
准教授 滝田 順子(たきた じゅんこ)
TEL:03-5800-8659(医局直通)FAX:03-3816-4108
E-mail:jtakita-tky“AT”umin.ac.jp
京都大学大学院医学研究科 腫瘍生物学講座
教授 小川 誠司(おがわ せいし)
TEL:075-753-9285(直通)FAX:075-753-9282
E-mail:sogawa-tky“AT”umin.ac.jp
取材に関するお問い合わせ
東京大学医学部附属病院 パブリック・リレーションセンター
担当:渡部、小岩井
TEL:03-5800-9188(直通)
E-mail:pr“AT”adm.h.u-tokyo.ac.jp
京都大学 企画・情報部広報課 国際広報室
担当:菊地
TEL:075-753-5729(直通)FAX:075-753-2094
E-mail:comms“AT”mail2.adm.kyoto-u.ac.jp
AMED事業に関するお問い合わせ
国立研究開発法人日本医療研究開発機構(AMED)
戦略推進部 がん研究課
次世代がん医療創生研究事業担当
〒100-0004 東京都千代田区大手町1-7-1
TEL:03-6870-2221 FAX:03-6870-2244
E-mail:cancer“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
関連リンク
掲載日 平成29年7月4日
最終更新日 平成29年7月4日


