プレスリリース 細菌感染症の診断に応用可能な迅速なゲノム解析システムの開発に成功―細菌種の同定時間を大幅削減。ポータブル化により災害現場や感染症多発地域での活用にも期待―
プレスリリース
東海大学
国立研究開発法人日本医療研究開発機構
東海大学(所在地:神奈川県伊勢原市下糟屋143、学長:山田 清志〔やまだ きよし〕)医学部基礎医学系 教授 今西 規〔いまにし ただし〕を中心とする研究グループは、ポータブル型のDNAシーケンサ「MinION」(Oxford Nanopore Technology社製)に応用可能なゲノム解析システムを開発し、16S rRNA遺伝子をPCR増幅して配列決定することにより、細菌感染症試料に対する細菌の同定を2時間以内で行うことが可能であることを示しました 。
従来から特定の細菌を検出するシステムはありましたが、このほど開発に成功した本システムは、ゲノム配列データベースに登録されている8万以上の細菌種・系統を特定できる能力を有している点が、従来のシステムとの大きな違いと言えます。
本研究は、国立研究開発法人日本医療研究開発機構(AMED)の感染症研究国際展開戦略プログラム(J-GRID)等の支援により行われました。
なお、本研究成果は平成29年7月18日(火)午前10時(イギリス時間)、オンラインジャーナル『Scientific Reports』(DOI:10.1038/s41598-017-05772-5)に掲載されました。
本研究成果のポイント
- 多くの医療機関では細菌感染症の原因菌判定には細菌培養法が使われており、結果が出るまでに1日から2日を要している
- 東海大学では、ナノポア技術を使った新型のDNA塩基配列決定装置と独自開発のソフトウエア群により、ポータブル型のゲノム解析システムを作製した
- 実際に感染症患者由来の試料を解析したところ、DNA抽出後から2時間以内に試料に含まれる細菌を同定できることを示した
研究の背景
一方で、医療機関における細菌感染症の診断は、多くの場合に細菌培養法によって行われていますが、培養法による細菌種の判定には1日から2日を要し、さらには難培養性細菌の存在や培養条件の不適合などの要因が菌種同定をしばしば困難にします。そうした理由から、細菌感染症が疑われる患者には原因菌の特定を待たずに経験的に選択された抗菌薬を投与する場合が多く、これが有効な薬剤投与の遅れや薬剤耐性菌出現の促進をもたらしていると考えられています。
研究の概要
 【図1】本研究で開発したポータブル型の細菌ゲノム解析システム。2台のラップトップ型PCと小型DNAシーケンサ「MinION」で構成される。
【図1】本研究で開発したポータブル型の細菌ゲノム解析システム。2台のラップトップ型PCと小型DNAシーケンサ「MinION」で構成される。
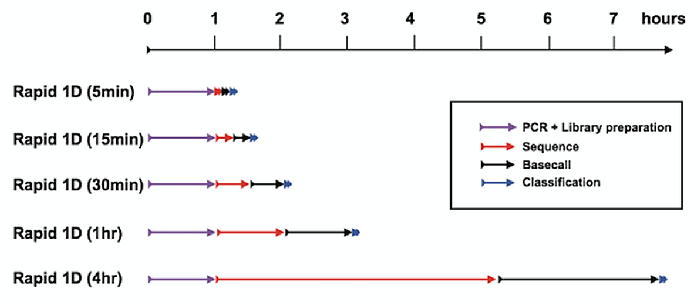 【図2】細菌種同定に要する時間の推定。Rapid 1D(5min)という方法を採用すると、PCR反応に約1時間と、その後シーケンスの実行に5分、その後PCでの解析を含めて、全体で2時間以内に判定結果を出すことができる。
【図2】細菌種同定に要する時間の推定。Rapid 1D(5min)という方法を採用すると、PCR反応に約1時間と、その後シーケンスの実行に5分、その後PCでの解析を含めて、全体で2時間以内に判定結果を出すことができる。
今後の展開
発表論文
†: equal contribution, *: corresponding author
研究予算
本研究は以下の研究予算の支援を受けて実施されました。
- 国立研究開発法人 日本医療研究開発機構(AMED)感染症研究国際展開戦略プログラム(J-GRID)の支援による研究課題「迅速・正確な感染症診断を可能にする病原微生物同定システムの開発」(代表:今西 規、平成27-29年度)
- 公益財団法人 大川情報通信基金 平成27年度研究助成の支援による研究課題「迅速な感染症診断のためのシーケンサ連動型ゲノム検索ソフトウエアの開発」(代表:今西 規)
- 文部科学省の私立大学戦略的研究基盤形成支援事業による東海大学の研究プロジェクト「高分子超薄膜から創成する次世代医用技術」(代表:喜多 理王、分担者:中川 草)
用語解説
- ▼DNAシーケンサ:
- 生物の遺伝情報を担うDNA(デオキシリボ核酸)の塩基配列を決定する装置
- ▼ナノポア:
- ナノメートル(10億分の1メートル)サイズの微小な穴
- ▼MinION(みないおん):
- Oxford Nanopore Technology社のDNAシーケンサの名称。ナノポアにDNA分子を通し、その際の電位変化に基づいて塩基配列を決定できる
- ▼細菌感染症:
- 細菌の感染によって引き起こされる感染症。黄色ブドウ球菌、ボツリヌス菌など、さまざまな菌が原因となる
- ▼薬剤耐性菌:
- 細菌感染症の治療に使われる抗菌薬に耐性を持つ細菌のこと。特に、複数の抗菌薬に対する耐性を持つ多剤耐性菌の出現は、世界的に深刻な問題となっている
- ▼相同性検索手法:
- 既知のDNA塩基配列に関するデータベースの中で、質問配列と一致する、または類似する塩基配列を検索するための計算手法。相同性検索の代表的なソフトウエアがBLASTである
お問い合わせ先
本件に関するお問い合わせ
東海大学医学部基礎医学系分子生命科学
担当:今西 規
TEL:0463-93-1121(代表)
事業に関するお問い合わせ
国立研究開発法人日本医療研究開発機構
戦略推進部 感染症研究課
TEL:03-6870-2225
E-mail:kansen“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
関連リンク
掲載日 平成29年7月20日
最終更新日 平成29年7月20日


