プレスリリース 細胞内におけるタンパク質―DNA相互作用の全体像を捉える新しい方法を開発
プレスリリース
国立大学法人九州大学
国立研究開発法人日本医療研究開発機構
九州大学大学院医学研究院医化学分野の梅山大地学術研究員(現:理化学研究所)と伊藤隆司教授は、細胞内におけるタンパク質-DNA相互作用の全体像を捉える新しい方法を開発しました。
私たちの身体を形成している様々な細胞は、基本的に同一のゲノムDNAを持っていますが、ゲノム中の遺伝子を取捨選択して使うことによって、それぞれの個性を発揮したり環境変化に適応したりしています。この取捨選択を行うのがDNAに結合する転写因子(※1)やヒストン(※2)等のタンパク質です。したがって、ゲノムの働き方を包括的に理解するには、ゲノムDNA上のタンパク質結合部位を網羅的に明らかにする必要があります。そのために、細胞から単離した核にDNA切断酵素を働かせる方法が用いられています。しかし、これらの方法は、操作が煩雑な上に、核を単離する過程でDNAとタンパク質の相互作用が失われる危険性も有しています。
これに対して、梅山博士と伊藤教授は、ジメチル硫酸(DMS)という細胞膜を通過してDNAをメチル化する化合物に着目しました。DMSを作用させた細胞からDNAを取り出して、メチル化部位で切断する反応を施してから次世代シーケンサ(※3)で分析すると、タンパク質の結合部位が切断を免れた場所として同定されました。DMS-seqと命名されたこの方法によって、核を単離せずに細胞内におけるタンパク質-DNA相互作用の全体像を明らかにすることが、初めて可能になりました。また、DNAを核内に収納する染色体の基本構造であるヌクレオソーム(※4)の中心位置を遺伝子操作なしに同定することにも初めて成功しました。DMS-seqは、様々な分野の研究を基礎から支える技術になることが期待されます。
本成果は、国立研究開発法人日本医療研究開発機構(AMED)の革新的先端研究開発支援事業ユニットタイプ「エピゲノム研究に基づく診断・治療へ向けた新技術の創出」研究開発領域における研究課題「ヒト消化器上皮細胞の標準エピゲノム解析と解析技術開発」(研究開発代表者:金井弥栄)および文部科学省科学研究費補助金によるもので2017年10月3日(火)12時(米国東部標準時[夏時間])に、国際学術雑誌「Cell Reports」のオンライン版に掲載されました。
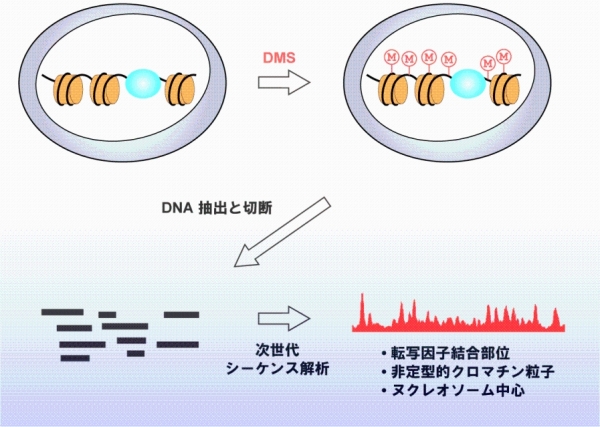 (参考図)
(参考図)
細胞に添加されたDMSは細胞膜を通過して、核内のDNAをメチル化しますが、タンパク質の結合部位はメチル化を免れます。DMSを作用させた細胞から抽出したDNAに、メチル化部位を切断する反応を加えてから次世代シーケンサで解析すると、DNA上のタンパク質の相互作用部位を包括的に明らかにできます。転写因子の結合部位のみならず、非定型的クロマチン粒子やヌクレオソームの中心も検出されます。
- 研究者からひとこと
- 生命科学の様々な分野における基礎的研究を支えるような独自の基盤技術や方法論の開発を心がけてゆきたいと思います。
背景
内容
そこで我々は、出芽酵母をモデルにDMS-seqの開発に取り組みました。その結果、期待通りに転写因子の結合部位を検出できることが判りました。例えば、転写因子Abf1が結合しそうな配列(結合モチーフ)の周辺にはin vivoでDMS-seqのピークが検出され(図1A)、DMSフットプリントとして検出されたものはChIP-seqピークとして検出されたものとよく一致しました(図1B)。また、転写の開始点と終結点の近傍に存在することが最近注目されている非定型的なクロマチン粒子も検出できました。更に、定型的なクロマチン粒子であるヌクレオソームについては、その中心座標に対してDMSが高親和性を示すという予想外の発見がありました(図1C)。ヌクレオソームの両端近傍を検出するMNase-seqとは切断点の分布が完全に逆位相を示します。従来、ヌクレオソームの中心座標を直接的に決定するにはヒストン遺伝子群の改変に基づく化学的切断法しか手段がなく、この方法が適用できる生物はヒストン遺伝子の数が例外的に少ない生物に限定されていました。これに対して、DMS-seqは、遺伝子操作を必要としませんので、原理的にはヒトを含むあらゆる真核生物においてヌクレオソームの中心座標の決定に応用可能な初めての方法となりました。
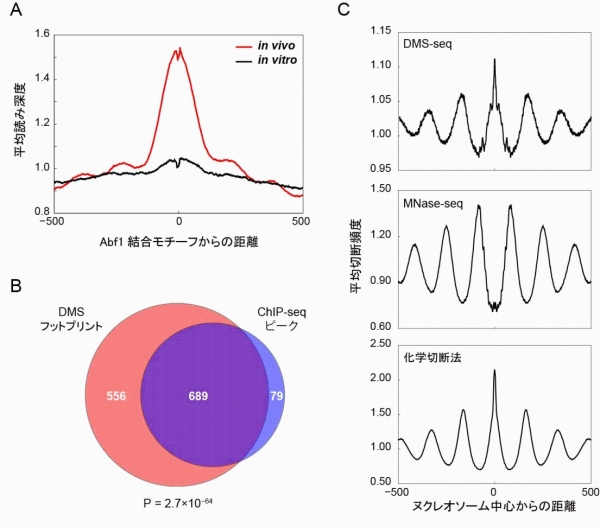 図1.DMS-seqによる転写因子結合とヌクレオソーム中心の検出
図1.DMS-seqによる転写因子結合とヌクレオソーム中心の検出
B. DMS-seqとChIP-seqの結果はよく一致する。DMSフットプリントとして検出されたAbf1結合モチーフはChIP-seqピークとして検出されたものと有意に重なる。
C. DMSはヌクレオソーム中心を優先的に切断する。DMS-seqによる切断点の分布は、ヌクレオソーム中心を検出する化学切断法による切断点の分布と一致する一方、ヌクレオソームの両端周辺を切断する酵素マイクロコッカルヌクレアーゼ(MNase)による切断点の分布とはちょうど逆位相の関係を示す。
今後の展開
本研究について
用語解説
- (※1)転写因子:
- DNAからRNAへの転写(遺伝子発現)を調節するタンパク質
- (※2)ヒストン:
- 長大なゲノムDNA分子に結合して核内への収納を助ける小型の塩基性核タンパク質
- (※3)次世代シーケンサ:
- DNAの配列を超並列で大量に読み取る解析装置
- (※4)ヌクレオソーム:
- 真核生物の染色体構造の基本構成単位で、4種類のヒストンを2分子ずつ含むタンパク質複合体に約150塩基対のDNAが巻き付いた構造
- (※5)クロマチン免疫沈降シーケンス法(ChIP-seq):
- 染色体を細断片化した上で、目的とするタンパク質の抗体を利用してそのタンパク質を含む断片を濃縮(免疫沈降)し、そこに回収されたDNAの配列を次世代シーケンサで解析することによって、そのタンパク質の結合部位を網羅的に明らかにする方法
論文名
(生体内のタンパク質-DNA相互作用とヌクレオソーム中心をゲノムワイドにマッピングするDMS-seq法)
雑誌名:Cell Reports
お問い合わせ先
研究に関するお問い合わせ
大学院医学研究院 教授 伊藤 隆司
電話:092-642-6095 FAX:092-642-2103
E-mail:tito“AT”med.kyushu-u.ac.jp
報道に関するお問い合わせ
九州大学広報室
電話:092-802-2130 FAX:092-802-2139
E-mail:koho“AT”jimu.kyushu-u.ac.jp
事業に関するお問い合わせ
国立研究開発法人日本医療研究開発機構(AMED)
基盤研究事業部 研究企画課
電話:03-6870-2224
E-mail:kenkyuk-ask“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
掲載日 平成29年10月23日
最終更新日 平成29年10月23日


