機械学習と次世代シークエンス技術の活用により日本人集団の白血球の血液型を解明
プレスリリース
大阪大学
国立遺伝学研究所
金沢大学
理化学研究所
日本医療研究開発機構
研究成果のポイント
- 日本人集団1,120名を対象に、次世代シークエンス技術※1を駆使して白血球の血液型を決定するHLA遺伝子※2を解読
- 機械学習※3により、日本人集団の白血球の血液型が11パターンの組み合わせで構成されることを解明
- バイオバンク・ジャパン※4が構築した日本人集団17万人を対象としたフェノムワイド関連解析(PheWAS)※5で、白血球の血液型の個人差が、病気や量的形質を含む50以上の表現型※6に関わることを示した
概要
大阪大学 大学院医学系研究科の平田潤大学院生、岡田随象教授(遺伝統計学)らの研究グループは、次世代シークエンス技術と機械学習を用いて、日本人集団における白血球の血液型が11パターンで構成されており、その個人差が、病気や量的形質を含む50以上の表現型に関わっていることを明らかにしました。
ヒトの血液に含まれる白血球には血液型が存在し、ヒトゲノム上のHLA遺伝子の配列の個人差で決定されます。白血球の血液型は移植医療や個別化医療 ※7に際して重要ですが、HLA遺伝子構造が複雑で解読に専門技術が必要なことや高額な実験費用により、HLA遺伝子配列の詳細な個人差の解明は遅れていました。
岡田教授らの研究グループは、最先端のゲノム配列解読技術である次世代シークエンス技術を駆使して、日本人集団1,120名を対象に33のHLA遺伝子におけるゲノム配列を決定することに成功しました。得られたHLA遺伝子ゲノム配列情報に対して機械学習手法であるtSNE ※8を適用した結果、日本人集団の白血球の血液型を11パターンの組み合わせに分類可能なことが明らかになりました。これは、複雑なヒトゲノム情報の解釈を、機械学習手法を用いて実現した先進的な成功例と評価することができます。
さらに研究グループは、日本人集団17万人のゲノムデータを対象に、白血球の血液型をコンピューター上で高精度に推定することに成功しました。推定された血液型パターンに基づき、多彩な表現型との関連を調べるフェノムワイド関連解析を実施しました。その結果、50以上の表現型において、白血球の血液型が発症に関与していることが明らかになりました。
本研究成果により、日本人集団における白血球の血液型の全容が解明されました。機械学習による白血球の血液型の分類に成功したことは、生命科学研究における機械学習の画期的な応用例と考えられます。さらに、白血球の血液型を用いた個別化医療の実現に貢献するものと期待されます。
本研究成果は、英国科学誌「Nature Genetics」に、1月29日(火)午前1時(日本時間)に公開されます。
研究の背景
ヒトの血液には、赤血球、白血球、血小板といった血球細胞が含まれています。赤血球の血液型(A、B、O、AB型)がヒトゲノム配列上の特定の遺伝子(=ABO遺伝子)のゲノム配列の個人差で決まるのと同様に、白血球にも血液型が存在し、HLA遺伝子のゲノム配列の個人差で決定されることが知られています。白血球の血液型の個人差は免疫アレルギー疾患をはじめ多彩な表現型の発症に強いリスクを有することが知られており、個人のゲノム情報を医療に活用する個別化医療の先駆けとして社会実装が期待されています。また、骨髄移植やiPS細胞を用いた再生医療などの臓器移植を伴う医療行為においては、臓器の提供者(ドナー)と移植を受ける人(レシピエント)との間で白血球の血液型を合致させる必要があることが知られています。白血球の血液型の構成は人種間で大きく異なることから、日本人集団における白血球の血液型の構成の全容解明が強く望まれていました。
しかし、多数の種類のHLA遺伝子が存在し、各遺伝子が数十種類以上の配列パターンを有するため、白血球の血液型の組み合わせが膨大な数となってしまうことから、その全体像は明らかになっていませんでした。さらに、HLA遺伝子配列の構造が複雑で、解読に多額の実験費用と専門的なデータ解析技術が必要となることも、全容解明を遅らせる原因となっていました。
本研究グループではこれまでに、7個の主要なHLA遺伝子(=古典的HLA遺伝子)を対象に、日本人集団における白血球の血液型の構成を決定し、日本人集団に特異的な白血球の血液型が存在することを報告していました(Okada Y et al. Nat Genet 2015)。今回、HLA遺伝子解析やゲノム情報解析を進めてきた研究者との共同研究により、HLA遺伝子の構成や病気との関連を詳細に検討しました。
本研究の成果
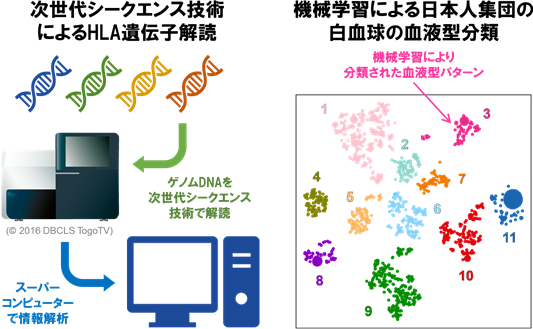
岡田教授らは、国立遺伝学研究所(井ノ上逸朗教授)および金沢大学医薬保健研究域医学系(細道一善准教授)との共同研究で、最先端のゲノム配列解読技術である次世代シークエンス技術を駆使することで、日本人集団1,120名を対象に33のHLA遺伝子における720種類のゲノム配列を詳細に決定することに成功しました(図1 左)。これまでに解明が進んでいなかった非古典的HLA遺伝子も解析対象に含まれているなど、日本人集団におけるHLA遺伝子配列のデータベースとして、最大級の情報を含んだ成果になります。
これらのゲノム配列の膨大な組み合わせを効率的に分類する目的で、得られたHLA遺伝子ゲノム配列情報に対して機械学習手法の一つであるt-SNEを適用しました。その結果、日本人集団の白血球の血液型を11パターンの組み合わせに分類可能なことが明らかになりました(図1 右)。これは、複雑なヒトゲノム情報の解釈を、機械学習手法を用いて実現した先進的な成功例と評価することができます。
さらに本研究グループは、理化学研究所生命医科学研究センター(鎌谷洋一郎チームリーダー)との共同研究で、1,120名で構築した白血球の血液型のパターン情報を学習データとして用いることで、バイオバンク・ジャパンが構築した日本人集団17万人における大規模ゲノムワイド関連解析(GWAS)※9のゲノムデータを対象に、白血球の血液型をスーパーコンピューター上で高精度に推定することに成功しました。得られた17万人の白血球の血液型パターンに基づき、病気(免疫疾患・生活習慣病・悪性腫瘍、etc.)や量的形質(身長・肥満、血液検査値、生理検査結果、etc.)を含む100を超える多彩な表現型との関連を網羅的に調べる、フェノムワイド関連解析を実施しました(図2)。本解析は、アジア人集団で実施されたフェノムワイド関連解析として、サンプル数および表現型数において過去最大規模の解析となります。その結果、これまで報告されていた免疫アレルギー疾患だけでなく、半数を超える52の表現型において、白血球の血液型が発症に関与していることが明らかになりました。これは、今まで想定されていたより広範囲の表現型の発症に、白血球の血液型の個人差が密接に関わっていることを示した結果と考えられます。

本研究成果が社会に与える影響(本研究成果の意義)
本研究成果により、日本人集団における白血球の血液型の全容が明らかとなり、移植医療への応用が期待されます。機械学習による白血球の血液型の分類に成功したことから、複雑なヒトゲノム情報に対する機械学習の応用研究におけるマイルストーンとなることが期待されます。さらに、白血球の血液型を用いて個人の表現型を予測し適切な医療を施す個別化医療の実現に貢献するものと期待されます。
用語説明
- ※1 次世代シークエンス技術
- 生物のゲノムを構成するDNA配列を高速に解読する技術。従来のゲノム解読手法であるサンガー法と比較して桁違いのスループットを誇り、幅広い生命科学研究における重要なツールとなっている。
- ※2 HLA遺伝子(human leukocyte antigen gene)
- ヒトの血球細胞の一種である白血球の表面に発現する分子で、白血球の血液型を規定する。生体内における自己と非自己の認識や外来性の病原菌に対する免疫反応を司り、多彩な表現型の個人差を規定している。主要な古典的HLA遺伝子(classical HLA gene)においては生物学的な役割の研究や検査方法の開発が進んでいるが、その他の非古典的HLA遺伝子(non-classical HLA gene)については解明が遅れている。
- ※3 機械学習(machine learning)
- コンピューターに高次元データを学習させることで、データの内部に潜む特徴的なパターンを見つけ出すデータ解析手法の総称。人工知能(artificial intelligence)研究におけるデータ解析手法としても広く活用されている。
- ※4 バイオバンク・ジャパン
- 日本人集団27万人を対象とした生体試料バイオバンクで、東京大学医科学研究所内に設置されている。ゲノムDNAや血清サンプルを臨床情報と共に収集し、研究者へのデータ提供や分譲を行っている。
- ※5 フェノムワイド関連解析(phenome-wide association study; PheWAS)
- 特定の遺伝子変異に着目し、多数の表現型との関連を網羅的に検討する解析手法。
- ※6 表現型
- 生物の外見や特徴として表現された形態的・生理的性質。代表的なヒトの表現型として、病気や身体的特徴(身長・肥満)、血液検査結果、生理検査結果などが含まれる。
- ※7 個別化医療
- 画一的な標準医療でなく、ヒトゲノム情報の違いなど患者さん一人一人の個性を考慮して施す次世代の医療。
- ※8 tSNE(t-distributed stochastic neighbor embedding)
- 機械学習手法の一つ。高次元データの次元を効率的に圧縮することで、低次元のデータ(例:2次元の画像データ)に変換する方法。
- ※9 ゲノムワイド関連解析(genome-wide association study; GWAS)
- ヒトゲノム配列上の数百万カ所の遺伝子変異と特定の表現型との関係を網羅的に検討する解析手法。数千人~百万人を対象に大規模に実施され、多数の表現型に対する遺伝子変異が同定されている。
研究者のコメント
- 岡田随象 教授
- HLA遺伝子の配列の個人差の解明は、移植医療の推進や個別化医療の実現など、日本人集団におけるゲノム情報の社会実装に不可欠と考えられています。本研究は、これまでHLA遺伝子を巡る疾患ゲノム研究を主導してきた私たちのチームと、長年にわたって日本人集団のHLA遺伝子解読技術開発に取り組んできた国立遺伝学研究所および金沢大学、日本を代表するゲノムコホートであるバイオバンク・ジャパンのゲノム情報解析を担ってきた理化学研究所が、共同研究を行うことにより達成することができました。すべての共同研究者や研究支援機構、サンプルを提供してくださった方々に感謝を申し上げます。
掲載論文
本研究成果は、2019年1月29日(火)午前1時(日本時間)に英国科学誌「Nature Genetics」(オンライン)に掲載されます。
- 【タイトル】
- “Genetic and phenotypic landscape of the MHC region in the Japanese population.”
- 【著者名】
- Jun Hirata1,2, Kazuyoshi Hosomichi3, Saori Sakaue1,4,5, Masahiro Kanai1,4,6, Hirofumi Nakaoka7, Kazuyoshi Ishigaki4, Ken Suzuki1,4,8, Masato Akiyama4,9, Toshihiro Kishikawa1,10, Kotaro Ogawa1,11, Tatsuo Masuda1,12, Kenichi Yamamoto1,13, Makoto Hirata14, Koichi Matsuda15, Yukihide Momozawa16, Ituro Inoue7, Michiaki Kubo17, Yoichiro Kamatani4,18, Yukinori Okada1,4,19,*. (* 責任著者)
- 【所属】
- 1大阪大学 大学院医学系研究科 遺伝統計学
2 帝人ファーマ株式会社 創薬探索研究所
3 金沢大学 医薬保健研究域医学系 革新ゲノム情報学分野
4 理化学研究所 生命医科学研究センター 統計解析研究チーム
5 東京大学 大学院医学研究科 アレルギー・リウマチ学
6 ハーバード大学メディカルスクール Department of Biomedical Informatics
7 情報・システム研究機構 国立遺伝学研究所 人類遺伝研究部門
8 東京大学 大学院医学研究科 糖尿病・代謝内科
9 九州大学 大学院医学研究院 眼科学
10 大阪大学 大学院医学系研究科 耳鼻咽喉科・頭頸部外科学
11 大阪大学 大学院医学系研究科 神経内科学
12 大阪大学 大学院医学系研究科 産科学婦人科学
13 大阪大学 大学院医学系研究科 小児科学
14 東京大学 医科学研究所シークエンス技術開発分野
15 東京大学 新領域創成科学研究科 メディカル情報生命専攻
16 理化学研究所生命医科学研究センター 基盤技術開発研究チーム
17 理化学研究所 統合生命医科学研究センター(研究当時)
18 京都大学・マギル大学ゲノム医学国際連携専攻
19 大阪大学 免疫学フロンティア研究センター(IFReC) 免疫統計学
特記事項
本研究は、日本医療研究開発機構(AMED)が支援するゲノム研究バイオバンク事業「オーダーメイド医療の実現プログラム(疾患関連遺伝子等の探索を効率化するための遺伝子多型情報の高度化)」の一環として行われ、文部科学省が推進する新学術領域研究「ゲノム科学の総合的推進に向けた大規模ゲノム情報生産・高度情報解析支援(ゲノム支援)」および「がんの複雑性のシステム的理解を目指した新次元の統合的研究(システム癌新次元)」、大阪大学先導的学際研究機構 生命医科学融合フロンティア研究部門、大阪大学大学院医学系研究科 バイオインフォマティクスイニシアティブの協力を得て行われました。
本件に関する問い合わせ先
研究に関すること
岡田随象(おかだ ゆきのり)
大阪大学 大学院医学系研究科 遺伝統計学 教授
(理化学研究所 生命医科学研究センター 統計解析研究チーム 客員主管研究員)
TEL:06-6879-3971 FAX:06-6879-3975
E-mail:yokada"AT"sg.med.osaka-u.ac.jp
報道に関すること
大阪大学大学院医学系研究科 広報室
TEL:06-6879-3388 FAX:06-6879-3399
Email:medpr"AT"office.med.osaka-u.ac.jp
国立遺伝学研究所 広報チーム 報道担当
TEL:055-981-5873 FAX:055-981-9418
E-mail:infokoho"AT"nig.ac.jp
金沢大学 総務部 広報室 広報係
TEL:076-264-5024 FAX:076-234-4015
E-mail:koho"AT"adm.kanazawa-u.ac.jp
理化学研究所 広報室 報道担当
TEL: 048-467-9272 FAX:048-462-4715
E-mail:ex-press"AT"riken.jp
AMED事業に関すること
国立研究開発法人日本医療研究開発機構
基盤研究事業部 バイオバンク課
〒100-0004 東京都千代田区大手町一丁目7番1号 読売新聞ビル
TEL:03-6870- 2228 FAX: 03-6870-2246
E-mail:order-made”AT”amed.go.jp
※E-mailは上記アドレス"AT"の部分を@に変えてください。
関連リンク
掲載日 平成31年1月29日
最終更新日 平成31年1月29日


