深層学習を用いて高精度にHLA遺伝子配列の予測が可能に―ヒトゲノム情報に対する深層学習の応用研究のマイルストーンに―
プレスリリース
大阪大学
東京大学
日本医療研究開発機構
研究成果のポイント
- 深層学習(ディープラーニング)※1を用いた新規のHLA imputation法※2、DEEP*HLAを開発し、これまで困難であった集団中に低頻度に存在するHLA(human leukocyte antigen)遺伝子※3配列の予測精度が上昇
- DEEP*HLAを1型糖尿病※4患者のゲノムデータに適用し、日本人集団と欧米人集団とで共通した発症リスクを有するHLA遺伝子配列を解明
- 大規模ヒトゲノム情報解析における深層学習の有用性を示すマイルストーン
概要
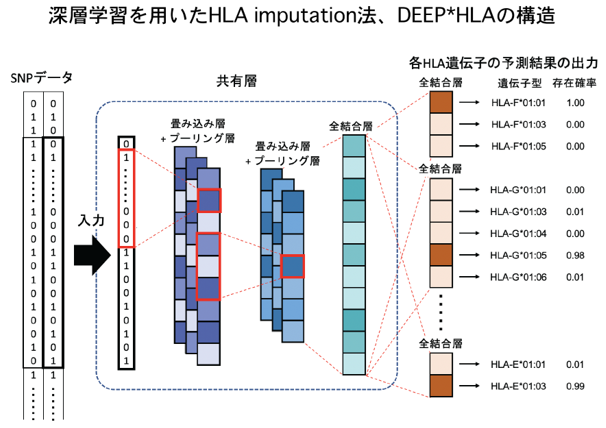
大阪大学大学院医学系研究科の特別研究学生の内藤龍彦さん(東京大学大学院医学系研究科博士課程)、岡田随象教授(遺伝統計学)らの研究グループは、東京大学大学院新領域創成科学研究科松田浩一教授らと共同で、免疫機能に関わるHLA遺伝子配列をコンピューター上で推測する、HLA imputation法に深層学習を応用した新たな手法、DEEP*HLAを開発しました(図1)。
HLA遺伝子は、白血球の血液型を規定し多彩な疾患に関わる重要な遺伝子です。個人におけるHLA遺伝子配列の解読において、多額の実験費用が掛かってしまうことが課題となっていました。そこで、HLA imputation法はHLA遺伝子配列をコンピューター上で予測する有効な手法として用いられています。しかし、従来のHLA imputation法では、集団中に低頻度で存在する特定のHLA遺伝子配列の予測精度が著しく低下してしまうという難点がありました。そのため、異なった人種集団間の統合解析など、様々なHLA遺伝子解析における解析結果の信頼性が低下してしまうという問題がありました。
今回、岡田教授らの研究グループが開発したDEEP*HLAという手法では、特に頻度が1%以下の稀なHLA遺伝子配列の予測において、従来の手法では予測精度60~70%台であったのが、約80%まで上昇しました。さらに、DEEP*HLAを1型糖尿病患者のゲノムデータに適用し、日本人と欧米人で共有されたリスクを持つHLA遺伝子配列の組み合わせを同定しました。この研究により、様々な疾患の発症と関連するHLA遺伝子配列の解明がさらに進むとともに、大規模ヒトゲノム情報に対する深層学習の応用研究のマイルストーンになることが期待されます。
本研究成果は、英国科学誌「Nature Communications」に、2021年3月12日(金)午後7時(日本時間)に公開されます。
研究の背景
ヒトの血液に含まれる血球細胞の1つである白血球は、自己と非自己を認識し外来性の抗原に対する攻撃を行う免疫反応を担う細胞です。赤血球の血液型と同様に、白血球にも血液型が存在し、HLA遺伝子のゲノム配列の個人差で決定されることが知られています。HLA遺伝子は、自己免疫疾患をはじめとした多彩な疾患の発症リスクと関連し、病態において重要な位置を占めることが知られています。HLA遺伝子配列は複雑で、解読に高いコストを要するため、ゲノム配列上で周囲に位置する複数の一塩基多型(SNP)※5の情報からHLA遺伝子配列をコンピューター上で網羅的に予測(HLA imputation法)して解析を行います。HLA imputation法により、様々な疾患の発症リスクに関連するHLA遺伝子配列が解明されましたが、報告されているリスク配列が人種間で異なるということがしばしば見られました。例えば1型糖尿病において、欧米人では、HLA-DQβ1分子の57番目がアスパラギン酸以外のアミノ酸であることが最も強く関連するリスク配列として知られていましたが、日本人では、同様の関連は認められないことが報告されていました。異なった人種集団間で統合解析を行うことによりさらなる知見が得られることが期待されますが、従来のHLA imputation法では低頻度のHLA遺伝子配列に対する予測精度が著しく低下してしまうため、低頻度配列を対象とする必要のある集団間の統合解析において信頼性の高い結果を得がたいという難点がありました。
このような従来の解析手法では困難な課題に対して、近年では機械学習※6手法をはじめとした人工知能(AI)技術※7に注目が集まっています。研究グループはその中でも、深層学習という手法に着目しました。深層学習は、多層ニューラルネットワークを用いた機械学習手法であり、主に画像認識や自然言語処理などの分野で既存手法を遥かに上回る予測性能を示すことから、2010年代中頃から現在までも注目されている技術です。一般的な統計学・機械学習の解析手法では捉えられないような複雑な特徴を学習できるという特性があります。
本研究の成果
研究グループは、深層学習を利用した新規のHLA imputation法、DEEP*HLAを開発しました(図1)。DEEP*HLAは、深層学習の中でも畳み込みニューラルネットワーク※8というモデルを用いており、教師データとなるHLA imputation用の参照用リファレンスデータを元に、SNP情報とHLA遺伝子配列の複雑な相関関係を学習します。学習したモデルは、SNP情報を入力として、HLA遺伝子配列の予測値を高精度で出力することができます。さらに、DEEP*HLAは、マルチタスク学習※9という深層学習手法を用いて、複数のHLA遺伝子の遺伝子配列の予測を同時に行うことで、精度の改善や実行時間の短縮を図りました。
日本人集団、欧米人集団の参照用リファレンスデータを用いて交差検証法により精度評価を行ったところ、DEEP*HLAは、従来の手法に比べて、特に頻度の低いHLA遺伝子配列の予測精度が改善していることがわかりました(図2、上)。また、新規手法は、従来法に比べて実行時間が短く、コンピューターの計算負荷の観点からも効率的であり、数十万人規模のビッグデータにも適用可能であることが示されました。
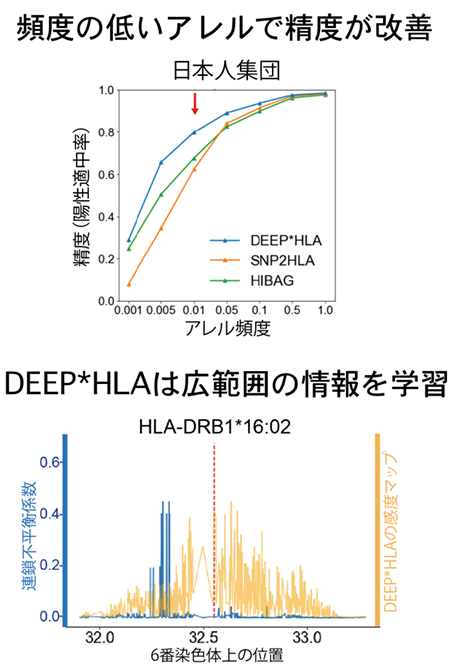
研究グループは、DEEP*HLAが入力情報であるHLA遺伝子周辺領域のどの部分を用いて学習予測を行っているかを調べるために、モデルの学習情報を可視化したところ、DEEP*HLAは近傍に位置する少数のSNP情報のみでなく、より広い領域に跨って分布する多数のSNP情報を用いて予測を行っているという結果が得られました(図2、下)。従来の手法は、近傍のSNP情報を用いた学習には強いのに対して、離れた箇所に位置するSNP情報を活用した学習を苦手としていましたが、DEEP*HLAはその点を克服したために予測精度が上昇した可能性が考えられました。
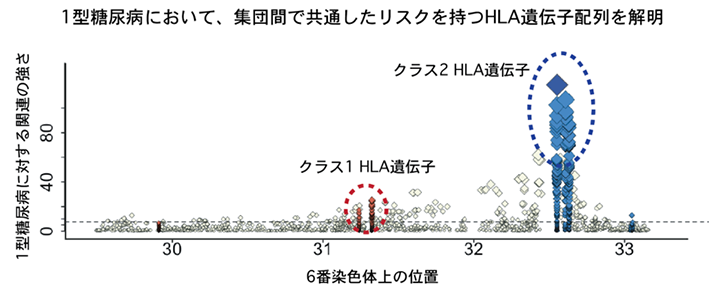
さらに研究グループは、1型糖尿病の発症に関わるHLA遺伝子配列を同定するために、バイオバンク・ジャパン※10が保有する日本人集団約6万人とUKバイオバンク※11が保有する欧米人集団約35万人のゲノム情報にDEEP*HLAを適用することで、HLA遺伝子配列を網羅的に推定しました。集団間で統合解析を行った結果、研究グループは、日本人と欧米人で1型糖尿病に対して共通したリスクを持つ複数のHLA遺伝子配列の組み合わせ(クラスⅠおよびクラスⅡ HLA遺伝子群)を同定することに成功しました(図3)。それらのHLA遺伝子配列は、いずれもHLA分子において機能的に重要な位置にあり、HLA分子の機能を介して1型糖尿病の発症に密接に関わっていると考えられます。
本研究成果が社会に与える影響(本研究成果の意義)
HLA遺伝子は多彩な疾患における発症に関わっており、病態の解明や個別化医療の実現の観点から、重要な領域として注目されています。今回、HLA遺伝子配列の予測精度の上昇により、異人種集団間の統合解析をはじめとしたHLA解析の信頼性が上がり、疾患と関連するHLA遺伝子配列の同定や病態のさらなる解明に大きく貢献できると期待されます。さらに、ヒトゲノム情報解析において深層学習が有用であることを示すマイルストーンとなり、同様の応用研究が加速すると期待されます。
特記事項
本研究成果は、2021年3月12日(金)午後7時(日本時間)に英国科学誌「Nature Communications」(オンライン)に掲載されます。
- タイトル
- “A deep learning method for HLA imputation and trans-ethnic MHC fine-mapping of type 1 diabetes”
- 著者名
- Tatsuhiko Naito1、2、Ken Suzuki1、Jun Hirata1、3、Yoichiro Kamatani4、Koichi Matsuda5、Tatsushi Toda2、and Yukinori Okada1、6、7*(* 責任著者)
- 所属
-
- 大阪大学大学院医学系研究科 遺伝統計学
- 東京大学大学院医学系研究科 神経内科学
- 帝人ファーマ株式会社 創薬探索研究所
- 東京大学大学院新領域創成科学研究科 メディカル情報生命専攻 複雑形質ゲノム解析分野
- 東京大学大学院新領域創成科学研究科 メディカル情報生命専攻 クリニカルシークエンス分野
- 大阪大学先導的学際研究機構 生命医科学融合フロンティア研究部門
- 大阪大学免疫学フロンティア研究センター 免疫統計学
なお、本研究は、日本医療研究開発機構(AMED)ゲノム医療実現推進プラットフォーム事業「次世代ゲノミクス研究による乾癬の疾患病態解明・個別化医療・創薬」の一環として行われ、大阪大学大学院医学系研究科バイオインフォマティクスイニシアティブの協力を得て行われました。
用語説明
- ※1 深層学習(ディープラーニング)
- 入力・中間・出力層の階層的なネットワーク構造(多層ニューラルネットワーク)を用いた機械学習手法の一種。他の機械学習手法では、捉えられないような複雑な特徴を直接抽出して学習できるという特性がある。
- ※2 HLA imputation法
- HLA遺伝子配列は、直接配列決定をするのに高いコストがかかるため、周囲のSNP情報との相関関係からコンピューターを用いて統計学的に推測すること。
- ※3 HLA(human leukocyte antigen)遺伝子
- 白血球の表面に発現する分子で、白血球の血液型を規定する。生体内における自己と非自己の認識や外来性の抗原に対する免疫反応に関わる。
- ※4 1型糖尿病
- 自己の免疫細胞が、インスリンを分泌性する膵臓β細胞を誤って攻撃してしまうことにより、インスリンの分泌が不足して血糖が上昇することにより起こる、糖尿病。
- ※5 一塩基多型(SNP)
- ヒトゲノムを構成している塩基配列の個体差で、配列上の一カ所が変化して生じるもの。
- ※6 機械学習
- 人工知能の一種で、大量のデータを学習することで、分類・予測や特徴抽出を行うコンピューターアルゴリズム。
- ※7 人工知能(AI)技術
- 人間が行う複雑なタスクや知的な振る舞いを、模倣もしくは超越するコンピューターシステム。
- ※8 畳み込みニューラルネットワーク
- 深層学習のモデルの一つで、特に画像の分類で高い予測能を発揮することで知られている。畳み込み層とプーリング層を用いて、画像の特徴量を直接抽出して学習することができる。
- ※9 マルチタスク学習
- 機械学習手法において、複数の異なる予測項目(タスク)を同時に学習して予測すること。上手く活用することで、実行時間の短縮や精度の上昇が期待される。
- ※10 バイオバンク・ジャパン
- 日本人集団約27万人を対象とした生体試料バイオバンクで、東京大学医科学研究所内に設置されている。ゲノムDNAや血清サンプルを臨床情報と共に収集し、研究者へのデータの公開や分譲を行っている。
- ※11 UKバイオバンク
- 英国の約50万人を対象とした国家的な生体試料バイオバンク。ゲノム情報や多彩な臨床情報、追跡情報を収集し、世界中の研究者にデータの公開や分譲を行っている。
本件に関するお問い合わせ先
研究に関すること
岡田随象(おかだゆきのり)
大阪大学 大学院医学系研究科 遺伝統計学 教授
TEL:06-6879-3971 FAX:06-6879-3975
E-mail:yokada“AT”sg.osaka-u.ac.jp
報道に関すること
大阪大学大学院医学系研究科 広報室
TEL:06-6879-3388 FAX:06-6879-3399
E-mail:medpr“AT”office.med.osaka-u.ac.jp
東京大学大学院新領域創成科学研究科 広報室
TEL:04-7136-5450
E-mail:press“AT”k.u-tokyo.ac.jp
AMED事業に関すること
国立研究開発法人日本医療研究開発機構
ゲノム・データ基盤事業部 ゲノム医療基盤研究開発課
ゲノム医療実現推進プラットフォーム事業 (先端ゲノム研究開発:GRIFIN)
TEL:03-6870-2228 FAX:03-6870-2243
E-mail:genome-platform“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
関連リンク
掲載日 令和3年3月15日
最終更新日 令和3年3月15日


