アジア人集団における遺伝的多型がマイクロRNA発現に及ぼす影響のカタログを作成―多彩なヒト疾患の発症に関連するマイクロRNAを同定―
プレスリリース
大阪大学
日本医療研究開発機構
研究成果のポイント
- アジア人集団における遺伝的多型※1がマイクロRNA※2発現に及ぼす影響を網羅的にカタログ化した。
- 個人の遺伝的多型情報からマイクロRNA発現量を予測する機械学習※3モデルを作成し、数十万人規模の疾患ゲノム情報に適用することで、多彩なヒト疾患の発症に関連するマイクロRNAを同定した。
- 本研究の成果を適用することで、アジア人における遺伝的疾患リスク関連領域の機能的意義の理解が進み、免疫アレルギー疾患を含む様々な疾患において新規治療標的の同定や病態解明につながることが期待される。
概要
大阪大学大学院医学系研究科の曽根原究人大学院生(遺伝統計学)、坂上沙央里助教(研究当時、現ハーバード大学医学部博士研究員)、岡田随象教授(遺伝統計学)らの研究グループは、アジア人集団における遺伝的多型がマイクロRNA発現に及ぼす影響のカタログを作成しました。そして、このデータを既報のゲノムワイド関連解析(GWAS: genome-wide association study)※4データと組み合わせることで、多彩なヒト疾患の発症に関連するマイクロRNA(miR-1908-5p)の同定に成功しました。
これまでの研究により、マイクロRNAは免疫アレルギー疾患を含む幅広い疾患の病態に関与することが示唆されてきました。また、これまで多数の疾患に対してGWASが実施され、疾患発症リスクと関連する多くの遺伝的多型(疾患感受性多型)が同定されてきました。一方で、個々の疾患感受性多型が疾患リスクに影響するメカニズムをマイクロRNAの観点から解明するためには、マイクロRNA発現に影響を及ぼす遺伝的多型(miRNA-eQTL※5)のカタログが必要となります。たとえば、関節リウマチや全身性エリテマトーデスといった免疫疾患への関与が示唆されているマイクロRNA miR-146aでは、その前駆体配列上に存在する遺伝的多型に応じて発現量が変化することが報告されています。これまでmiRNA-eQTLに関する研究は主に欧米人を対象としてなされており、ゲノム構造の違いを考慮すると、アジア人集団で見られる疾患感受性多型の詳細な解明のためには、アジア人集団を対象としたmiRNA-eQTL研究が必要とされていました。
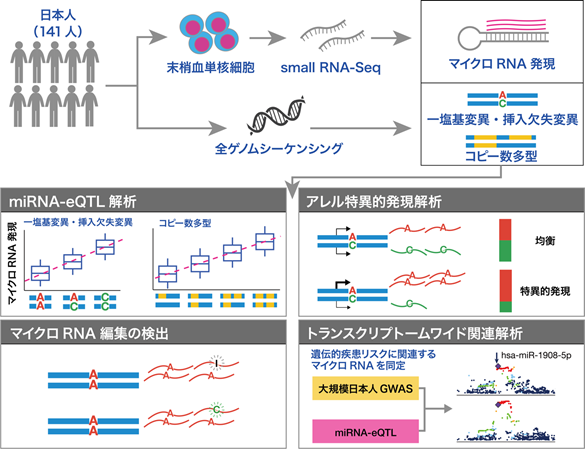
今回、研究グループは、日本人141名から採取した末梢血単核細胞※6中のマイクロRNA発現とゲノム配列情報との関連を網羅的に解析する(図1)ことで、アジア人集団におけるmiRNA-eQTLのカタログの作成に成功しました。さらに、構築したカタログを日本人を対象とした既報のGWASデータと組み合わせることにより、疾患発症に関与するマイクロRNAをスクリーニングするトランスクリプトームワイド関連解析(TWAS: transcriptome-wide association study)※7を実施しました。
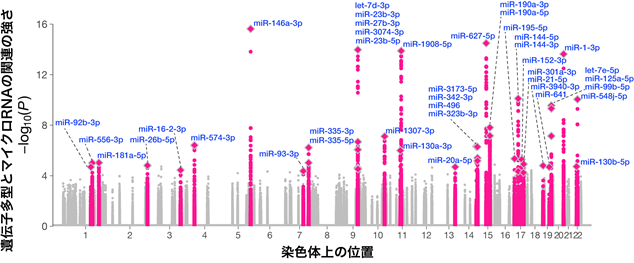
その結果、マイクロRNA miR-1908-5pが、多彩なヒト疾患の発症リスクと関連することがわかりました。本研究成果は、マイクロRNAを介した遺伝的疾患リスクの解明を通じ、免疫アレルギー疾患を含む様々な疾患において新規治療標的の同定や病態解明、診断技術の開発につながることが期待されます。
本研究成果は、英国科学誌「Human Molecular Genetics」に2022年2月1日(火)午前0時(日本時間)に公開されました。
研究の背景
ゲノムワイド関連解析(GWAS)では、疾患発症リスクと関連を示す疾患感受性多型を同定することで、疾患発症の原因解明の手がかりを得ることができます。これまで数多くのGWASがなされ、数万にも及ぶ疾患感受性多型が同定されてきました。同定された疾患感受性多型の多くが、ゲノム上のタンパク質を直接コードする領域の外に位置することが分かっており、その機能的意義の理解が課題となっています。この課題に対する有望なアプローチとして、eQTL解析があります。eQTL解析では遺伝子発現量に影響する遺伝的多型をカタログ化することで、疾患感受性多型を遺伝子発現量の変化の観点から理解することを可能とします。
一方で、ノンコーティングRNA※8の一種であるマイクロRNAは免疫疾患、がん、代謝性疾患といった幅広い疾患の病態への関与が示唆されてきました。このため、マイクロRNAのeQTL(miRNA-eQTL)情報を用いることで、GWASで同定された疾患感受性多型をマイクロRNA発現量の個人差の観点から理解することが期待されます。しかし、既報のmiRNA-eQTL研究は数が限られている上に、その多くが欧米人を対象としています。欧米人とアジア人のゲノム構造の違いを考慮すると、欧米人集団における研究で得られたmiRNA-eQTL情報を、アジア人集団で見出された疾患感受性多型に適用することには困難があり、アジア人集団を対象としたmiRNA-eQTL研究が必要とされていました。
本研究の成果
今回、研究グループは、日本人集団141名の末梢血単核細胞分画からRNAを抽出し、small RNA-Seq※9によりマイクロRNA発現量を網羅的に定量しました。さらに全ゲノムシーケンシング※10により得られたゲノム配列情報と、マイクロRNA発現量との関連解析を実施することで、日本人集団におけるmiRNA-eQTLのカタログを作成しました(図2)。今回の解析で検出したmiRNA-eQTLの中には、欧米人集団においては遺伝的多型自体が存在しないものも含まれており、その発見はアジア人を対象とした研究を実施したことによる成果といえます。
従来のmiRNA-eQTL研究では、個人におけるマイクロRNA発現の総量と遺伝子多型情報との関連を検定する手法が主として用いられてきました。本研究ではそれに加えて、small RNA-Seqで得られたリード中に含まれる遺伝的多型情報を活用することで、マイクロRNAのアレル特異的発現(ASE: allele-specific expression)※11の解析を実施しました。これにより、従来の関連解析では検出されなかった、遺伝的多型の影響を受けるマイクロRNA(miR-933)の同定に成功しました。
また、従来のmiRNA-eQTL研究では、解析対象の遺伝的多型として一塩基変異(SNV: single-nucleotide variant)※12および小規模な挿入欠失変異(short InDel: short insertion and deletion)※13のみが考慮されてきました。本研究ではそれに加えて、全ゲノムシーケンシングデータを基にゲノム全体の大規模なコピー数多型領域(copy number variation: CNV)※14を決定することで、マイクロRNA発現とCNVとの関連を解析しました。その結果、CNVの影響を受けるマイクロRNA(miR-570-3p)の同定に成功しました。
さらに、得られたマイクロRNA発現量とゲノム配列情報を統合することで、個人の遺伝的多型情報を基にマイクロRNA発現量を予測する機械学習モデルを構築しました。このモデルを既報の日本人集団における数十万人規模の疾患GWAS情報に適用することで、遺伝的に制御されたマイクロRNA発現量と疾患リスクとの関連を調査するTWASを実施しました。その結果、マイクロRNA miR-1908-5pの減少が、大腸がんおよび2型糖尿病を含む多彩なヒト疾患の罹患リスクと関連することがわかりました。このことは、miR-1908-5pがこれらの疾患の発症機序に関与していることを示唆し、診断バイオマーカー※15や創薬ターゲットとしての活用が期待されます。
本研究成果が社会に与える影響(本研究成果の意義)
本研究において作成されたmiRNA-eQTLカタログを利用することで、アジア人集団における疾患感受性多型の背後に潜む分子メカニズムの解明が加速することが期待されます。
また、本研究で同定された疾患関連マイクロRNA miR-1908-5pについては、大腸がんおよび2型糖尿病の罹患リスク判定に利用可能なバイオマーカーとしての応用が期待されます。今後、培養実験や動物実験といったさらなる検証を進めていくことで、これらの疾患の創薬標的となることも期待されます。
特記事項
本研究成果は、2022年2月1日(火)午前0時(日本時間)に英国科学誌「Human Molecular Genetics」(オンライン)に掲載されました。
- タイトル
- “Genetic architecture of microRNA expression and its link to complex diseases in the Japanese population”
- 著者名
- Kyuto Sonehara1,2,18, Saori Sakaue1,3–6,18*, Yuichi Maeda2,7,8, Jun Hirata1,9, Toshihiro Kishikawa1,10,11, Kenichi Yamamoto1,12,13, Hidetoshi Matsuoka14, Maiko Yoshimura14, Takuro Nii7,8, Shiro Ohshima14, Atsushi Kumanogoh2,7,15, Yukinori Okada1,2,13,16,17*
(* 責任著者) - 所属
-
- 大阪大学大学院医学系研究科 遺伝統計学
- 大阪大学先導的学際研究機構 生命医科学融合フロンティア研究部門
- ハーバード大学医学部 Center for Data Sciences
- ブリガム&ウィメンズ病院 Divisions of Genetics and Rheumatology
- ブロード研究所 Program in Medical and Population Genetics
- 理化学研究所 生命医科学研究センター 統計解析研究チーム
- 大阪大学大学院医学系研究科 呼吸器・免疫内科学
- 大阪大学大学院医学系研究科 免疫制御学
- 帝人ファーマ株式会社 創薬探索研究所
- 大阪大学大学院医学系研究科 耳鼻咽喉科・頭頸部外科学
- 愛知県がんセンター 頭頸部外科部
- 大阪大学大学院医学系研究科 小児科学
- 大阪大学 免疫学フロンティア研究センター 免疫統計学
- 大阪南医療センター リウマチ・膠原病・ アレルギー科
- 大阪大学 免疫学フロンティア研究センター 感染病態分野
- 大阪大学 感染症総合教育研究拠点
- 理化学研究所 生命医科学研究センター システム遺伝学チーム
- 共同筆頭著者
本研究は、日本医療研究開発機構(AMED)免疫アレルギー疾患実用化研究事業の採択課題「疾患ゲノム情報を活用した自己免疫疾患における核酸ゲノム創薬の推進」、「免疫オミクス情報の横断的統合による関節リウマチのゲノム個別化医療の実現」(研究開発代表者:岡田随象)の一環として行われ、大阪大学先導的学際研究機構、大阪大学大学院医学系研究科 バイオインフォマティクスイニシアティブの協力を得て行われました。
用語説明
- ※1 遺伝的多型
- ゲノムを構成するDNAの塩基配列のうち、個体間で異なる部位。特に疾患発症リスクと関連する部位を、疾患感受性多型と呼ぶ。
- ※2 マイクロRNA
- 遺伝子の転写後発現調節に関与する、21~25塩基程度の短鎖ノンコーティングRNA。ヒトゲノムにはおよそ2000種のマイクロRNAが存在することが知られている。マイクロRNAは標的遺伝子のmRNAに結合し、結合したmRNAの安定性の変化やタンパク質への翻訳の阻害を介して遺伝子発現を調節する。
- ※3 機械学習
- 与えられたデータを元に、その背景にあるルールやパターンを学習するコンピューターアルゴリズムの総称。
- ※4 ゲノムワイド関連解析(GWAS: genome-wide association study)
- 遺伝的多型と形質(疾患の有無などを含む、個々人の性質や特徴)との関連を、ゲノム全域にわたって網羅的に探索する解析。現在の一般的なGWASでは、ゲノム全域で数百~数千万に及ぶ遺伝的多型が解析に用いられる。
- ※5 eQTL
- expression quantitative trait loci。遺伝子発現量の個人差と関連するゲノム領域。
- ※6 末梢血単核細胞
- 末梢血から分離された単核細胞成分。単球やリンパ球といった免疫細胞から構成される。
- ※7 トランスクリプトームワイド関連解析(TWAS: transcriptome-wide association study)
- ゲノム上の遺伝的多型情報を元に、遺伝的に制御された遺伝子発現量をゲノム全域にわたり予測し、その予測値と形質との関連を網羅的に探索する解析。
- ※8 ノンコーディングRNA
- DNAから転写された後に、タンパク質へ翻訳されずに生理的機能を示すRNAの総称。
- ※9 small RNA-Seq
- 次世代シーケンサーと呼ばれる、一度の実験で大量の塩基配列情報を読み出すことができる装置を用いることで、マイクロRNAを含む、15~150塩基程度の小分子RNAの発現を網羅的に解析する手法。
- ※10 全ゲノムシーケンシング
- 次世代シーケンサーを用いることで、個人の全ゲノム配列を網羅的に解析する手法。
- ※11 アレル特異的発現(ASE: allele-specific expression)
- ヒトを含む2倍体の生物のゲノムは、その各領域において父親由来・母親由来の2つからなる一対の遺伝子を有する。これら2つの遺伝子間で遺伝子の発現量に不均衡があるとき、アレル特異的発現と呼ぶ。
- ※12 一塩基変異(SNV: single-nucleotide variant)
- ゲノムDNAの塩基配列中の1塩基が異なる塩基に変化している現象。
- ※13 挿入欠失変異(InDel: insertion and deletion)
- ゲノムDNAの塩基配列中において、何らかの塩基配列の挿入または欠失が生じている現象。ここではshort InDelとして、50塩基以下のものを指す。
- ※14 コピー数多型領域(CNV: copy number variation)
- 1細胞あたりのコピー数が個人間で異なるゲノム上の領域。
- ※15 診断バイオマーカー
- ある疾患の有無や進行状況、治療効果を定量的に把握するための指標。生体中のタンパク質や遺伝子が代表的なものとして挙げられる。
本件に関するお問い合わせ先
研究に関すること
岡田 随象(おかだ ゆきのり)
大阪大学 大学院医学系研究科 遺伝統計学 教授
TEL:06-6879-3971 FAX:06-6879-3975
E-mail:yokada"AT"sg.med.osaka-u.ac.jp
報道に関すること
大阪大学大学院医学系研究科 広報室
TEL:06-6879-3388 FAX:06-6879-3399
E-mail:medpr”AT”office.med.osaka-u.ac.jp
AMED事業に関すること
日本医療研究開発機構(AMED)
疾患基礎研究事業部 疾患基礎研究課
免疫アレルギー疾患実用化研究事業
TEL:03-6870-2286
E-mail:nambyo-i”AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
関連リンク
掲載日 令和4年2月3日
最終更新日 令和4年2月3日


