DNAの傷を修復するタンパクを呼び込む新たなメカニズムを解明
成果情報
横浜市立大学
日本医療研究開発機構
横浜市立大学大学院生命医科学研究科 奥田昌彦特任准教授と西村善文学長補佐は、名古屋大学環境医学研究所 発生遺伝分野 荻 朋男教授、長崎大学 原爆後障害医療研究所 中沢由華助教らとの共同研究で、遺伝子情報を読み出す転写の途中で、障害となるDNAの傷を修復するためにUVSSAタンパク質がTFIIHタンパク質を呼び込む姿を、NMR分光器を用いて観察し、そのメカニズムを解明しました。修復が正常に働かないと、遺伝性の難病であるコケイン症や、紫外線高感受性症候群を発症することから、その修復過程の詳細情報は、当該疾患の発症プロセス解明や治療薬開発に役立つと考えられます。
研究成果のポイント
- 転写に伴ってDNAを修復する新たな機構の解明に繋がった
- ゲノム全体でのDNA修復経路と転写に伴うDNA修復経路において、修復タンパク質を呼び込む機構が大変似ていることを発見した
- コケイン症や紫外線高感受性症候群など転写に伴うDNA修復の欠損により発症する疾患の治療薬の開発に貢献できる
研究の背景
私たちのDNAは、有害な代謝副産物、発癌物質、紫外線等の様々な要因により、常に損傷の危機に直面しています。重要な遺伝子が傷ついてしまうと病気を引き起こし、また傷が残ってしまうと誤った遺伝情報が次の世代に引き継がれてしまいます。しかしDNAの傷を完全に回避することはできません。この重大な問題に対して私たちの身体(細胞)は、完璧な防御策を講じるよりも、入ってしまった傷を修復する素晴らしいしくみを築き上げてきました。その一つが「ヌクレオチド除去修復」と呼ばれるしくみで、損傷部位と周辺のヌクレオチド(*1)を除いて新しいものに作り替えます。
ヌクレオチド除去修復において、XPCタンパクは、ゲノム(*2)全体に渡り、DNAに生じた傷を最初に見つけて修復を開始する働きをします。DNAの傷を見つけた後、XPC は損傷周辺のヌクレオチドを切断するために、DNAの二重らせんを解く基本転写因子(*3)TFIIHを呼び込みます。TFIIHは10種類の異なるタンパクからなる大きな複合体で、普段は転写を開始するために働いていますが、DNA修復でも重要な役割を果たします。
ヌクレオチド除去修復には、XPCによる開始経路の他に転写に伴う経路があります。この経路では、DNAを鋳型にしてRNAを合成しながら進んで行く途中に傷があると、そこで合成が止まってしまい、これが信号となって修復反応が開始されるのですが、多くのタンパクが関与する複雑な反応のため、詳しい機構は明らかになっていません。この経路ではどのタンパクがTFIIHを呼び込むのか、XPCによるTFIIHの呼び込みのしくみとの関連性についても長らく不明でした。
ヌクレオチド除去修復において、XPCタンパクは、ゲノム(*2)全体に渡り、DNAに生じた傷を最初に見つけて修復を開始する働きをします。DNAの傷を見つけた後、XPC は損傷周辺のヌクレオチドを切断するために、DNAの二重らせんを解く基本転写因子(*3)TFIIHを呼び込みます。TFIIHは10種類の異なるタンパクからなる大きな複合体で、普段は転写を開始するために働いていますが、DNA修復でも重要な役割を果たします。
ヌクレオチド除去修復には、XPCによる開始経路の他に転写に伴う経路があります。この経路では、DNAを鋳型にしてRNAを合成しながら進んで行く途中に傷があると、そこで合成が止まってしまい、これが信号となって修復反応が開始されるのですが、多くのタンパクが関与する複雑な反応のため、詳しい機構は明らかになっていません。この経路ではどのタンパクがTFIIHを呼び込むのか、XPCによるTFIIHの呼び込みのしくみとの関連性についても長らく不明でした。
研究の内容
本研究では、転写にともなって作用するヌクレオチド除去修復因子の1つUVSSAタンパクがTFIIH複合体を呼び込むために、TFIIHの構成メンバーであるp62タンパクのPHドメインと結合した立体構造を、横浜市立大学の高磁場NMR分光器(*4)を用いて決定しました(図1)。
これによりUVSSAは、普段は決まった構造を作らない領域でp62のPHドメインと強く結合することが分かりました。この結合では、アスパラギン酸やグルタミン酸といった酸性アミノ酸を多く含む約20アミノ酸ほどの短い領域が、伸びた紐のような構造を形成してp62のPHドメインの分子表面の多くの塩基性アミノ酸のリジンと静電的相互作用しながら巻き付いていました。また、酸性アミノ酸に囲まれたフェニルアラニンとバリンがp62のPHドメインのポケットに入り込むことが特異的な結合に重要であることが分かりました。これらのアミノ酸をアラニンに置換した変異UVSSAを細胞に導入すると、紫外線照射によって生じるDNAの傷をうまく修復出来なくなってしまい、RNAポリメラーゼが合成反応を再開する効率が大きく低下してしまいました(図2)。変異UVSSAがp62のPHドメインに結合できないためにTFIIHを損傷箇所に呼び寄せる際に問題が生じ、修復がうまく行われなくなってしまったことが原因であると考えられます。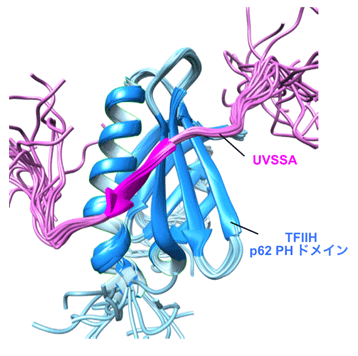
これによりUVSSAは、普段は決まった構造を作らない領域でp62のPHドメインと強く結合することが分かりました。この結合では、アスパラギン酸やグルタミン酸といった酸性アミノ酸を多く含む約20アミノ酸ほどの短い領域が、伸びた紐のような構造を形成してp62のPHドメインの分子表面の多くの塩基性アミノ酸のリジンと静電的相互作用しながら巻き付いていました。また、酸性アミノ酸に囲まれたフェニルアラニンとバリンがp62のPHドメインのポケットに入り込むことが特異的な結合に重要であることが分かりました。これらのアミノ酸をアラニンに置換した変異UVSSAを細胞に導入すると、紫外線照射によって生じるDNAの傷をうまく修復出来なくなってしまい、RNAポリメラーゼが合成反応を再開する効率が大きく低下してしまいました(図2)。変異UVSSAがp62のPHドメインに結合できないためにTFIIHを損傷箇所に呼び寄せる際に問題が生じ、修復がうまく行われなくなってしまったことが原因であると考えられます。
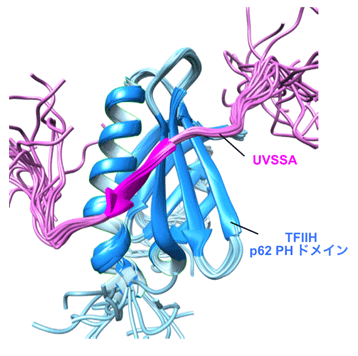
(図1)UVSSAがTFIIH p62のPHドメインを捉えた姿 (20個の構造を重ねて表示)
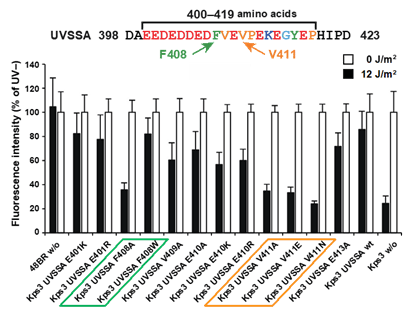

(図2)UVSSAがTFIIH p62のPHドメインに結合できなくなると、DNA損傷部位で停止したRNAポリメラーゼの転写反応の回復が低下することを細胞内で明らかにした実験
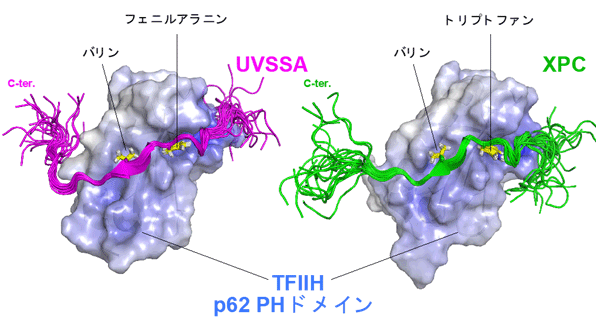
今回の研究で明らかになったUVSSAが同じp62のPHドメインを捕らえてTFIIHを呼び込むしくみは、XPCが使うしくみ(参考文献1)ととても類似していることが分かりました(図3)。このしくみは転写でも使われています。転写が開始される時にTFIIHを呼び込むのは、同じ基本転写因子であるTFIIEαや、p53やDP1などの転写活性化因子であり、これらのタンパクがそれぞれの酸性領域でp62のPHドメインを捕まえTFIIHを呼び込みます(参考文献2~4)。この時、これらのタンパクはXPCやUVSSAのように、普段構造を作らない酸性アミノ酸に富んだ領域で、伸びた紐のような構造でしかも、とても似たしくみでPHドメインを捕らえるのです。このことから、さまざまなタンパクが共通したルールに基づいて同じタンパクを認識していることが分かりました。

(図3)UVSSAがTFIIH p62のPHドメインを捉えた姿はXPCとよく似ている(p62 PHドメインは分子表面の電荷を表示。青色は正電荷を表している。UVSSAとXPCはリボンモデルで20個の構造を重ねて表示。)
今後の展開
ヌクレオチド除去修復は、UVSSA 、XPCやTFIIHの他にも多くのタンパクが関わる、多段階からなる複雑な過程です。これらのタンパクの遺伝子変異は、紫外線高感受性症候群、色素性乾皮症、コケイン症候群、硫黄欠乏性毛髪発育異常症などの遺伝性疾患を発症します。各段階においてそれぞれのタンパクがどのような役割を果たしているのかを一つ一つ明らかにすることが発症メカニズムの解明につながります。
今回の研究では、ヌクレオチド除去修復で知られている2つの経路のうち、転写に伴う方の経路におけるTFIIHのDNA損傷箇所への呼び込みをUVSSAが担っていることを明らかにし、それらの複合体の立体構造からそのしくみを解明しました。今後は、UVSSA自身がどのように損傷箇所に呼び込まれるのか、また呼び込まれたTFIIHがどのようにしてDNA二重らせんを解き、切断酵素とやりとりをするのか等のしくみを調べる必要があります。このような研究の成果は、新薬設計に有益な情報を提供することが期待されます。
今回の研究では、ヌクレオチド除去修復で知られている2つの経路のうち、転写に伴う方の経路におけるTFIIHのDNA損傷箇所への呼び込みをUVSSAが担っていることを明らかにし、それらの複合体の立体構造からそのしくみを解明しました。今後は、UVSSA自身がどのように損傷箇所に呼び込まれるのか、また呼び込まれたTFIIHがどのようにしてDNA二重らせんを解き、切断酵素とやりとりをするのか等のしくみを調べる必要があります。このような研究の成果は、新薬設計に有益な情報を提供することが期待されます。
参考文献
- (1)Masahiko Okuda, Minoru Kinoshita, Erina Kakumu, Kaoru Sugasawa, Yoshifumi Nishimura.
- “Structural insight into the mechanism of TFIIH recognition by the acidic string of the nucleotide excision repair factor XPC” Structure (2015) 23, 1827−1837
- (2)Masahiko Okuda, Aki Tanaka, Manami Satoh, Shoko Mizuta, Manabu Takazawa, Yoshiaki Ohkuma,Yoshifumi Nishimura
- “Structural insight into the TFIIE-TFIIH interaction: TFIIE and p53 share the binding region on TFIIH” The EMBO Journal (2008) 27, 1161−1171
- (3)Masahiko Okuda, Yoshifumi Nishimura
- “Extended string binding mode of the phosphorylated transactivation domain of tumor suppressor p53” Journal of the American Chemical Society (2014) 136, 14143−14152
- (4)Masahiko Okuda, Keigo Araki, Kiyoshi Ohtani, Yoshifumi Nishimura
- “The interaction mode of the acidic region of the cell cycle transcription factor DP1 with TFIIH” Journal of Molecular Biology (2016) 428, 4993−5006
掲載論文
- Common TFIIH recruitment mechanism in global genome and transcription-coupled repair subpathways
- Masahiko Okuda, Yuka Nakazawa, Chaowan Guo, Tomoo Ogi, Yoshifumi Nishimura
Nucleic Acids Research, gkx970, https://doi.org/10.1093/nar/gkx970
用語説明
- (*1)ヌクレオチド:
- 核酸(DNAやRNA)の構成単位
- (*2)ゲノム:
- 遺伝情報の総体。遺伝子(gene)と染色体(chromosome)を組み合わせた用語
- (*3)基本転写因子:
- RNAポリメラーゼIIが、タンパク質合成の鋳型であるメッセンジャーRNAの合成反応を開始するの を補助するタンパク。TFIIA、 TFIIB、TFIID、TFIIE、TFIIF、TFIIHの6タンパクをいう。
- (*4)NMR分光器:
- 強い磁場中で特定の原子核スピンの向きが揃えられた化合物やタンパク質等に対し、ラジオ波を照射して核磁気共鳴させた後、 核スピンが元の安定な状態に戻る際に出す信号を観測して、原子の配置などを解析する装置
お問い合わせ先
本資料の内容に関するお問合せ
横浜市立大学 大学院生命医科学研究科 奥田 昌彦
横浜市立大学 大学院生命医科学研究科 西村 善文
TEL:045-508-7211/7212
E-mail:okuda777“AT”yokohama-cu.ac.jp(奥田)
E-mail:nisimura“AT”yokohama-cu.ac.jp (西村)
取材対応窓口、資料請求など
横浜市立大学 研究企画・産学連携推進課長 渡邊 誠TEL:045-787-2510
E-mail:kenki“AT”yokohama-cu.ac.jp
AMED事業に関するお問い合わせ
国立研究開発法人日本医療研究開発機構(AMED)創薬戦略部 医薬品研究課
TEL:03-6870-2219
E-mail:20-DDLSG-16“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
関連リンク
掲載日 平成29年11月24日
最終更新日 平成29年11月24日


