成果情報 膜タンパク質の耐熱化変異体を合理的に設計―立体構造に基づく創薬を促進する―
成果情報
理化学研究所
千葉大学
日本医療研究開発機構
理化学研究所(理研)放射光科学研究センター理研RSC-リガク連携センターの国島直樹客員研究員、千葉大学大学院理学研究院の村田武士教授らの共同研究グループ※は、「膜タンパク質」分子の立体構造に基づく「耐熱化変異体」を計算により合理的に設計する新技術を開発しました。
本研究成果は、膜タンパク質の立体構造を決定するために必要な結晶試料の生産効率を向上させ、創薬応用を含む構造生物学[1]の発展に貢献すると期待できます。
構造に基づく創薬では、疾病関連タンパク質の原子レベルの立体構造をもとに候補化合物を設計します。創薬ターゲットの半数以上を占める膜タンパク質は、熱安定性が低いものが多く、立体構造を得るのに必要な質および量の結晶試料を生産することが困難です。膜タンパク質試料の生産効率を向上するためには、熱安定性を高めた耐熱化変異体が有効ですが、そのような変異体はこれまで非効率な試行錯誤により作出されてきました。
今回、共同研究グループは、膜タンパク質の膜外部分に変異導入する耐熱化設計を自由エネルギー計算[2]に基づいて合理的に行う技術を提案し、その有効性をモデルタンパク質の好熱性ロドプシン(TR)[3]によって確かめました。本技術を広く適用することで、構造解析が不可能な膜タンパク質の割合が減り、創薬応用を含む構造生物学の促進、ひいては将来の医療問題の解決につながると考えられます。
本研究は、科学雑誌『Proteins: Structure, Function, and Bioinformatics』のオンライン版(2020年10月16日付)に掲載されました。
- ※共同研究グループ
- 理化学研究所 放射光科学研究センター
リサーチアソシエイト(研究当時) 松浦祥悟(まつうらよしのり)
利用技術開拓研究部門 生体機構研究グループ
先任研究員 内藤久志(ないとうひさし)
理研RSC-リガク連携センター
客員研究員 国島直樹(くにしまなおき)
千葉大学 大学院 理学研究院
教授 村田武士(むらたたけし)
大学院融合理工学府
博士前期課程(研究当時) 秋山友希(あきやまともき)
博士前期課程2年 風間一輝(かざまかずき)
大学院理学研究科
博士前期課程(研究当時) 根本紗也加(ねもとさやか)
スペクトリス株式会社マルバーン・パナリティカル事業部
プロダクトスペシャリスト 廣瀬雅子(ひろせまさこ)
岡山大学 薬学部
教授 須藤雄気(すどうゆうき) - ※研究支援
- 本研究は日本医療研究開発機構(AMED)創薬等支援技術基盤プラットフォーム事業「SPring-8におけるワンストップタンパク質試料生産支援および高分解能結晶取得技術の高度化(研究代表者:国島直樹)」および日本学術振興会(JSPS)科学研究費補助金新学術領域研究(研究領域提案型)「発動分子を合理設計・理論計算するための物理化学評価と構造基盤の確立(研究代表者:村田武士)」、「ロドプシンを起動分子とした「化学・力学・光」エネルギー発動機構の理解と利用(研究代表者:須藤雄気)」による支援を受けて行われました。
背景
構造生物学研究の基盤情報であるタンパク質分子の原子分解能の立体構造は、目的タンパク質を結晶化し、その構造(結晶構造)を決定して得ることが一般的です。これまでに実施された日本の構造ゲノム科学[4]プロジェクトでは、ある耐熱性バクテリアが持つ全タンパク質の立体構造決定を目指して網羅的な結晶構造解析が行われましたが、解析可能な結晶が得られ、かつ立体構造の決定に成功したタンパク質は全体の約2割に過ぎませんでした。一つの生物において全体の約8割のタンパク質が結晶化できないという状況は、現在に至るまで解消されていません。
この状況に対処すべく、国島直樹客員研究員らはこれまでさまざまな技術開発に取り組んできました。例えば、部位特異的変異導入[5]により合理的に結晶化確率を高める方法注1)、X線自由電子レーザー[6]施設「SACLA[7]」を使って微小な多数の結晶から構造決定する方法注2)、合成鉱物を結晶化の足場として利用する新規結晶化法注3)などが挙げられます。
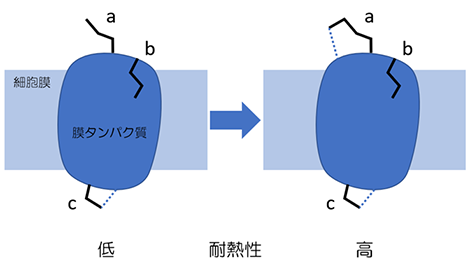
「膜タンパク質」は、構造生物学における最も重要な研究対象の一つですが、細胞膜に埋もれて存在するため一般的に熱安定性が低く、構造解析のための試料調製が特に困難とされています。この問題への対処法としては、熱安定性を高めた「耐熱化変異体」を取得して試料生産を向上させるものがあり、これにより膜タンパク質の立体構造決定に成功した例がいくつか報告されています。しかし、耐熱化変異体の取得は、これまで非効率的な試行錯誤により行われてきたため、合理的な方法の開発が望まれていました。
2015年、国島直樹客員研究員らは、大腸菌由来のCutA1という水溶性モデルタンパク質について、100℃以上の温度に耐える“超耐熱化”変異体を合理的に設計することに成功しました注4)。タンパク質の熱安定性は、結晶構造に基づく自由エネルギー計算によりある程度予測できます。CutA1では、この方法で超耐熱化変異体を設計し、その変性温度を86℃から137℃まで高めることができました。
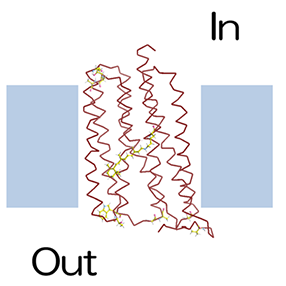
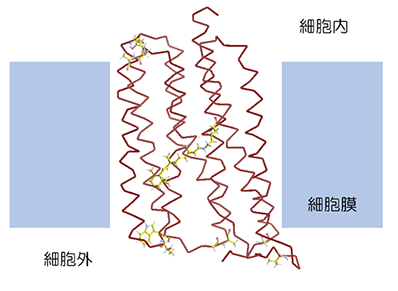
そこで、今回共同研究グループは、膜タンパク質の立体構造決定を効果的に支援するため、この耐熱化変異体設計法の膜タンパク質への適用を試みました。ただし、この方法はもともと水溶性タンパク質を想定しているため、膜タンパク質の膜外部分を設計対象としました(図1)。また、超耐熱化膜タンパク質の設計を目指すため、CutA1と同程度の耐熱性を持つ膜タンパク質である好熱性ロドプシン(TR)をモデルタンパク質として選びました(図2)。このTRの結晶構造は、今回の共同研究グループである千葉大および岡山大グループにより2016年に決定されています注5)。
注1)2018年7月30日理化学研究所プレスリリース「タンパク質の結晶化を実験的に診断」
注2)Naitow et al. (2017) "Protein-ligand complex structure from serial femtosecond crystallography using soaked thermolysin microcrystals and comparison with structures from synchrotron radiation" Acta Cryst. D73, 702-709
注3)Sugahara et al. (2008) "Nucleant-mediated protein crystallization with the application of microporous synthetic zeolite" Acta Cryst. D64, 686-695
注4)2015年10月26日理化学研究所プレスリリース「100℃以上の温度でのタンパク質の安定化機構を熱力学的に解明」
注5)Tsukamoto et al. (2016) "X-ray crystallographic structure of thermophilic rhodopsin: implications for high thermal stability and optogenetic function" J. Biol. Chem. 291, 12223-12232
研究手法と成果
共同研究グループは、①膜外領域の変異導入部位の選択、②変性状態の自由エネルギー計算、③変異による構造変化などの予測、④耐熱化変異候補の決定、という手順で膜タンパク質TRの耐熱化変異体を設計しました。この設計手順は、パソコンなど構造生物学の一般的な研究設備と膜タンパク質の結晶構造があれば短期間で実行できます。
まず、変異を導入する部位として、野生型TRの結晶構造において膜外領域に存在し溶媒に露出しているアミノ酸残基を28カ所選びました。結晶構造を観察することで選択しましたが、タンパク質構造の揺らぎを考慮する分子動力学(MD)シミュレーション[8]の結果も参考にしました。結晶構造では溶媒に露出していても、生体中では膜やタンパク質分子内部に埋もれる残基もあり、そのような可能性のある残基はMDシミュレーションにより除外しました。
次に、これら28残基それぞれについて、全種類のアミノ酸に置換した場合の変性自由エネルギー変化[2]を計算しました。この計算結果から、熱安定性が向上すると予測される20種類の単一変異について変異体モデルを作成し、その中から変異導入に伴って構造変化が起こると予測される変異体などを除きました。以上の手順で、最終的に10種類の耐熱化変異体候補を得ました。
設計された10種類のTR変異体について実際にタンパク質試料を調製し、それらの耐熱性をレチナール色素熱退色の速度論的測定[9]により実験的に評価しました。さらに、示差走査熱量測定(DSC)[10]によりタンパク質熱変性を分析しました。その結果、10種類の変異体のうち4種類に野生型に比べて耐熱化が認められました(図3)。設計の成功率は40%であり、これは以前に水溶性タンパク質CutA1で得た成功率39%と同等であることから、この設計法が膜タンパク質にも適用できることが示されました。
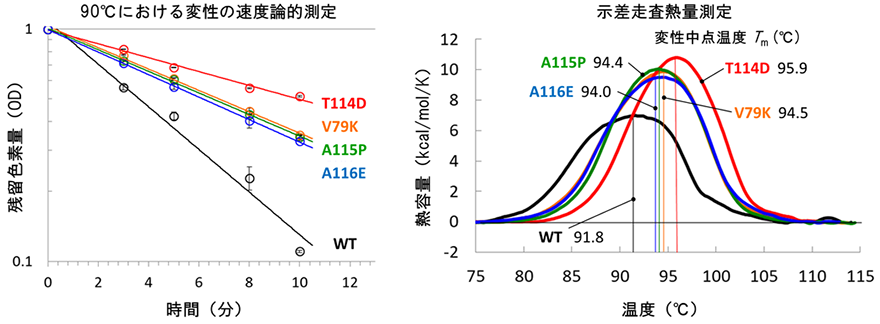
右:タンパク質熱変性の示差走査熱量測定の結果を示す。野生型(WT)に比べて、4種類の変異体(V79K、T114D、A115P、A116E)は高い変性中点温度を示し、より耐熱化したといえる。
(Akiyama et al. Proteins DOI:10.1002/prot.26015 Figure 2より改変)
さらに、耐熱化設計の結果を考察するため2種類(天然状態および変性状態)のMDシミュレーションを行いました。まず、天然状態のタンパク質の立体構造を検討するため、水溶液中に膜とTR分子を配置したモデルを使って、常温300K(27℃)で100ナノ秒(ns、1nsは10億分の1秒)のMDシミュレーションを行いました(図4左)。変異体のMD構造を確認したところ、変異導入部位はほぼ設計通りの構造になっており、例えば耐熱化に成功したV79K変異体(79番目のバリンをリジンに置換したもの)では、設計において意図した通りの静電気相互作用によるイオンペア[11]を確認しました(図4右)。さらに、MD構造と結晶構造の間の構造変化を統計的に分析した結果、10種類のうち9種類の変異体は野生型と同程度の構造変化を示し、分子全体で見る限りうまく設計できたと考えられます。しかし、TR分子内の局所的な構造変化パターンが変異型と野生型とで異なる場合も見られました。このことが耐熱化の成功率を下げている可能性があり、今後の検討課題となりました。
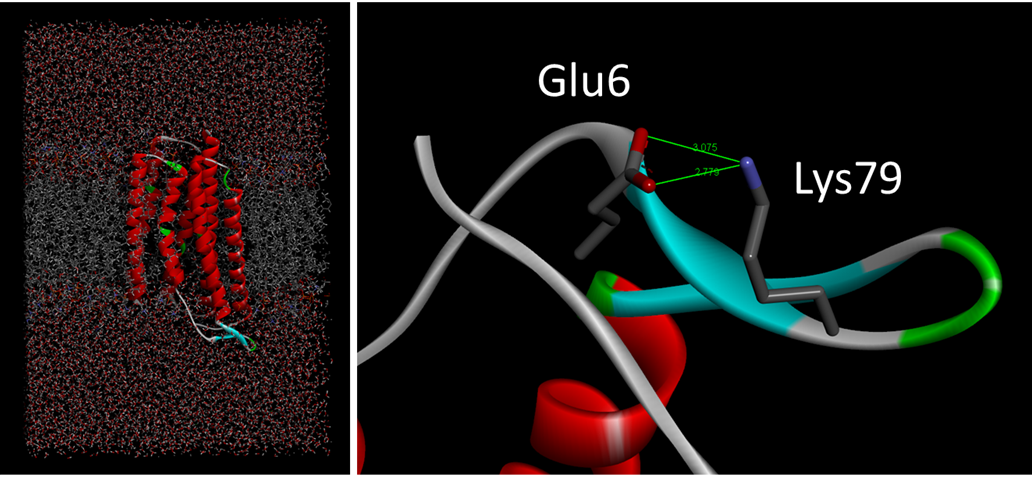
右:V79K変異体の変異導入部位周辺のMD構造。タンパク質のリボンモデル上に、棒状モデルで描いた6番目のグルタミン酸と79番目のリジンが、緑線で示す静電的相互作用によりイオンペアを形成している。
(Akiyama et al. Proteins DOI:10.1002/prot.26015 Figure 3より改変)
一方、耐熱化に成功したT114D変異体(114番目のスレオニンをアスパラギン酸に置換したもの)では、分子全体で見て野生型と比べて有意に大きな(MD−結晶構造間の)構造変化を示しました。このT114D変異体は変異導入により構造が変化し、タンパク質の機能に影響する可能性があります。この結果は、天然状態で大きな構造変化を起こす変異導入をMDシミュレーションにより予測できることを意味しています。この変異導入による構造変化のメカニズム解明も今後の課題です。
次に、変性状態におけるTR分子の立体構造を検討するため、高温1800K(1527℃)で1nsの短時間MDシミュレーションを行いました。タンパク質の変性状態の構造はよく分かっていませんが、決まった形に折り畳まれた天然状態の構造と比べて揺らぎが大きいほどけた構造で、その揺らぎの程度すなわちエントロピー(乱雑さを表す物理量)がタンパク質の熱安定性に関与することが知られています。本来であれば、実際の熱変性が起こる370K(97℃)付近の温度で長時間のMDシミュレーションを行うべきですが、現状では計算能力の制限などから実現できません。そこで、常温(300K)で平衡化した後、高温で1ns程度の短時間MDシミュレーションをすることで、変性状態に近い構造を作り出すことを試みました。300~2000K(27~1727℃)の範囲のさまざまな温度を検討した結果、1800Kにするとランダムコイル状態[12]と呼ばれる揺らぎの大きい状態の構造を再現性良く作り出せることが分かりました(図5)。

(Akiyama et al. Proteins DOI:10.1002/prot.26015 Figure 3より改変)
そこで、この高温短時間MDシミュレーションを20回繰り返し、TRのランダイムコイルMD構造を統計的に分析しました。その結果、耐熱化に失敗したS8D変異体(8番目のセリンをアスパラギン酸に置換したもの)のランダイムコイルMD構造は、他の変異体や野生型のそれに比べて有意に揺らぎが大きいことが分かりました。真の変性状態でもこの傾向が維持されているとすると、S8D変異の導入により変性状態でのエントロピーが増大したことで、変性の自由エネルギーが小さくなり、結果的に耐熱性が低下したと考えられます。この結果は、変性状態のエントロピーが増大する変異体をMDシミュレーションにより予測できることを意味しています。しかし、なぜS8D変異体で揺らぎが大きくなるのかは不明で、その解明は今後の課題です。
以上の結果から、本技術により、膜タンパク質の耐熱化変異体を約4割の成功率で合理的に設計できることが分かりました。今後、MDシミュレーションを併用することで、成功率はさらに向上する可能性があります。膜タンパク質の合理的な生産効率の向上が一歩前進したといえる成果です。
今後の期待
2050年に65歳以上の高齢者が4割近くを占めると予測される日本において、医療問題は避けて通ることのできない課題です。創薬ターゲットの半数以上は構造解析の難しい膜タンパク質と言われています。本研究成果の合理的設計技術を広く適用することで解析不可能なタンパク質の割合が減り、創薬応用を含む構造生物学の促進、ひいては将来の医療問題の解決につながると期待できます。
論文情報
- タイトル
- Further thermo-stabilization of thermophilic rhodopsin from Thermus thermophilus JL-18 through engineering in extramembrane regions
- 著者名
- Tomoki Akiyama, Naoki Kunishima, Sayaka Nemoto, Kazuki Kazama, Masako Hirose, Yuki Sudo, Yoshinori Matsuura, Hisashi Naitow, and Takeshi Murata
- 雑誌
- Proteins: Structure, Function, and Bioinformatics
- DOI
- 10.1002/prot.26015
補足説明
- [1]構造生物学
- 生物を構成する生体高分子、特にタンパク質や核酸の分子機能を、その立体構造から理解することを目指す生物学の一分野。X線結晶構造解析、核磁気共鳴、電子顕微鏡などの技術を用いる。
- [2]自由エネルギー計算、自由エネルギー変化
- タンパク質の熱変性を、決まった形に折り畳まれた構造を持つ天然状態からほどけた揺らぎの大きい構造を持つ変性状態への可逆的な2状態転移と捉えると、熱変性に伴うギブス自由エネルギー変化はΔG=ΔH-TΔSと表される(ΔHはエンタルピー変化、ΔSはエントロピー変化、Tは絶対温度)。タンパク質の結晶構造をもとに、このギブス自由エネルギー変化を見積もることが可能であり、これを「自由エネルギー計算」と呼んでいる。本研究では、TRの結晶構造(野生型)および結晶構造からモデル化した変異体構造をもとに、FoldXというプログラムを用いた変性の自由エネルギー計算により、野生型および変異体の耐熱性を予測した。
- [3]好熱性ロドプシン(TR)
- 米国にある約75℃の温泉で見いだされた高度好熱菌Thermus thermophilus JL-18が持つ耐熱性の膜タンパク質で、光駆動のプロトン(水素イオン)ポンプとして機能する。レチナールと呼ばれる色素および260残基のアミノ酸から成り、細胞膜を貫通する7本のヘリックスから成る膜内領域と二つの膜外領域を持つ。細胞内から細胞外への膜を横切るプロトンの能動的な輸送は、膜内領域に存在するレチナールが光を感じて構造変化し、それに連動してタンパク質分子が構造変化することで起こる。TRは耐熱性が高いことから、光によって動物の行動を制御するオプトジェネティクスなどに利用されている。TRはThermophilic Rhodopsinの略。
- [4]構造ゲノム科学
- 生体内のタンパク質の立体構造を機能未知なものも含めて網羅的に決定し、タンパク質の立体構造と機能の関係を総合的に研究することで生命現象を理解しようとする学問分野。
- [5]部位特異的変異導入
- 目的タンパク質の遺伝子に組換え技術などを利用して変異を導入し、当該タンパク質分子の任意の位置に任意のアミノ酸配列を導入する分子生物学の手法。例えば、タンパク質の機能を人為的に改変するために用いられる。
- [6]X線自由電子レーザー
- X線領域におけるレーザーのこと。従来の半導体や気体を発振媒体とするレーザーとは異なり、真空中を高速で移動する電子ビームを媒体とするため、原理的な波長の制限はない。また、数フェムト秒(1フェムト秒は1,000兆分の1秒)の超短パルスを出力する。
- [7]SACLA
- 理化学研究所と高輝度光科学研究センターが共同で建設した日本で初めてのX線自由電子レーザー施設。2011年3月に施設が完成し、SPring-8 Angstrom Compact free electron LAserの頭文字を取ってSACLAと命名された。2011年6月に最初のX線レーザーを発振、2012年3月から共用運転が開始され、利用実験が行われている。大きさが諸外国の同様の施設と比べて数分の1とコンパクトであるにもかかわらず、0.1ナノメートル(nm、100億分の1m)以下という世界最短波長のレーザーの生成能力を持つ。
- [8]分子動力学(MD)シミュレーション
- タンパク質などの水溶液中での揺らぎ、動的環境を調べるために用いられる。経験的な力場を用いて、古典力学におけるニュートン方程式を数値解析により解き、各原子の位置と速度の時間展開を得ることで、その系の動的過程を解析する。結晶構造は水溶液中で取り得る多数の異なる立体構造のうち、1構造のスナップショットに過ぎないが、MDではタンパク質構造の揺らぎを考慮することで、水溶液中の構造などをシミュレーションできる。MDはMolecular Dynamicsの略。
- [9]レチナール色素熱退色の速度論的測定
- ロドプシンの光センサーであるレチナール色素は、可視光領域の光を吸収する。加熱によりレチナールが不活性化すると、この光吸収が消失(熱退色)し、それに伴ってロドプシンの機能も失われる。本研究では、この現象を利用し、90℃における波長530nmの光吸収を時系列で測定することでTRの耐熱性を評価した。
- [10]示差走査熱量測定(DSC)
- 試料セルと参照セルを同時に一定速度で昇温させ、試料の構造変化を熱量の出入りから観測する。タンパク質変性の熱量を直接的に測定できるので、仮定を含まない熱力学量が得られる。本研究では、測定されたDSCカーブのピーク温度を変性中点温度(Tm)としてTRの耐熱性を評価した。DSCはDifferential Scanning Calorimetryの略。
- [11]イオンペア
- 互いに逆の電荷を持つ二つのイオンが静電的相互作用により結合したもの。静電的相互作用は遠距離力であり、5~6オングストローム(Å)程度離れた距離まで作用する。タンパク質では、イオン化する側鎖を持つアミノ酸の側鎖間などでイオンペアが生じる。タンパク質のイオンペアは、特に100℃以上の高温領域においてタンパク質の熱安定性に寄与することが知られている。
- [12]ランダムコイル状態
- タンパク質の立体構造状態の一種で、ヘリックスやシートなどの二次構造を持たず揺らぎの大きい状態。ランダムコイル構造は、タンパク質のペプチド鎖が完全に伸びた状態ではなく、分子内相互作用はある程度存在する。真の変性状態は二次構造が部分的に保たれたモルテングロビュール状態と呼ばれる状態に近いと考えられているが、真の変性状態のMDシミュレーションは計算時間の制限などから難しい。本研究では、高温の短時間MDによりランダムコイル状態を再現性良くシミュレーションできることを利用し、ランダムコイルMD構造を分析することでTRの変性状態を検討した。
発表者・機関窓口
発表者
理化学研究所 放射光科学研究センター 理研RSC-リガク連携センター
客員研究員 国島直樹(くにしまなおき)
千葉大学大学院 理学研究院
教授 村田武士(むらたたけし)
機関窓口
理化学研究所 広報室 報道担当
E-mail:ex-press“AT”riken.jp
*今般の新型コロナウイルス感染症対策として、理化学研究所では在宅勤務を実施しておりますので、メールにてお問い合わせ願います。
千葉大学 企画総務部 渉外企画課 広報室
E-mail:koho-hp“AT”office.chiba-u.jp
AMED事業に関する連絡先
日本医療研究開発機構(AMED) 創薬事業部 医薬品研究開発課
創薬等ライフサイエンス研究支援基盤事業(BINDS)
Tel:03-6870-2219 Fax:03-6870-2244
E-mail:20-ddlsg-16“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
関連リンク
掲載日 令和2年11月25日
最終更新日 令和2年11月25日


