成果情報 不要な細胞の除去機構の発見―核内因子が直接細胞膜に作用する!―
成果情報
京都大学アイセムス(物質-細胞統合システム拠点)
京都大学生命科学研究科
日本医療研究開発機構
- 生体内から不要な細胞を除去するメカニズムの発見
- 死にゆく細胞を用いて遺伝子同定する新しい方法の開発
- 核内タンパク質による細胞膜タンパク質の直接的な制御を明らかに
京都大学アイセムス(物質-細胞統合システム拠点)の鈴木淳教授、圓岡真宏特定助教、Daniel Packwood講師らの研究グループは、京都大学生命科学研究科の原田浩教授、徳島大学の小迫英尊教授と協力し、生体内において不要な細胞を除去する分子メカニズムを明らかにしました。この成果は、2021年3月15日に米国科学雑誌モレキュラーセル誌(セルプレス)にてオンライン公開され、2021年4月1日号に掲載されます。
概要
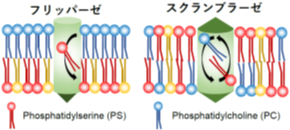
毎日、生体内では10~100億個の細胞がプログラムされた細胞死(アポトーシス)を起こしています。細胞死を起こした細胞が生体内に残存すると、細胞内容物が漏出し、これが免疫細胞を活性化させることで、炎症を引き起こしてしまいます。そのため、死んだ細胞は自分自身が適切に処理されるための目印を細胞表面に提示し、マクロファージ等の食細胞により認識され、貪食注1)により除去されています。死んだ細胞はスクランブラーゼ注2)というタンパク質を用いて細胞膜を構成する脂質の一つホスファチジルセリン(PS)を“死んだ目印”として細胞表面に提示しますが、スクランブラーゼがどのように活性化するのか、その詳細な機構は分かっていませんでした。
今回、アイセムスの研究グループは、ゲノム編集技術であるクリスパーCas9を用いたリバイバルスクリーニングという独自のスクリーニング法を開発し、スクランブラーゼの活性化因子を同定しました。驚くことに、この活性化因子は通常は核内に存在しDNA修復注3)に関与しており、カスパーゼとよばれる酵素によって切断されることでその一部が細胞質内に放出され、細胞膜中のスクランブラーゼに直接結合することでスクランブラーゼを活性化させることがわかりました。今回対象としたスクランブラーゼは脳の神経細胞で強く発現しており、脳内において不要な細胞の除去により神経変性疾患を防ぐなど脳の健康維持に関与していると考えられています。今後、見出された不要細胞除去システムを活用することで、脳内における不要細胞を除去し脳の健康を維持する新しい技術の開発が期待されます。
背景
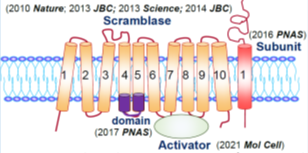
生体においては、細胞の増殖と死が厳密に制御されることで恒常性が維持されています。細胞死を起こした細胞は細胞膜の内側に存在しているリン脂質のホスファチジルセリン(PS)を”死んだ目印”として細胞表面に提示し、貪食細胞はPSに対する受容体によって死細胞表面のPSを認識し貪食しています。PSが”死んだ目印”として機能することは1980年代より示唆され、PSを細胞表面に露出させるスクランブラーゼの存在が仮定されていましたが、その分子的実体は長らく分かっていませんでした(図1)。鈴木教授らのグループはこれまで、世界に先駆けて、Xkrファミリー、TMEM16ファミリーといったPSを露出するスクランブラーゼのファミリーを同定し、その機能解析を進めてきました(2010 Nature, 2013 J Biol Chem, 2013 Science, 2014 J Biol Chem)。また、スクランブラーゼの細胞膜への局在に必要なサブユニット、活性に必要なドメインも解明しています(2016 PNAS, 2017 PNAS, 総説2021 Neurosci Res)。一方でスクランブラーゼがどのように活性化するのかに関しては未だ謎が多く残されていました。今回の研究では、これまで不明であったスクランブラーゼの活性化因子を同定しました(2021 Mol Cell)(図2)。これにより、死んだ細胞が細胞表面に“死んだ目印”を提示する精巧なシステムが明らかとなり、特に脳内において不要な神経細胞がどのように除去されるのか、そのメカニズムの一端が解明されました。
研究内容と成果
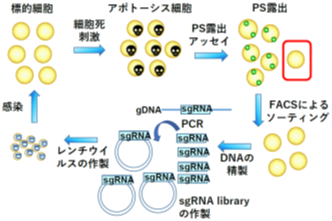
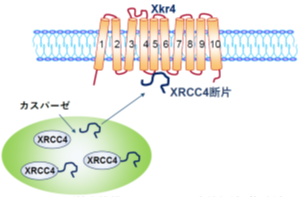
今回、研究グループはスクランブラーゼの中でも特に神経細胞に強く発現しているXkr4に注目しました。Xkr4はカスパーゼによって自身のC末端側が切断されることで2量体を形成しますが、それだけでは活性化せず、活性化因子の存在が仮定されていました。アポトーシスを起こし死にゆく細胞はDNAが分解され、細胞が増殖できないため、通常は遺伝子同定が極めて困難です。そのため、未知の活性化因子の同定には、死にゆく細胞を用いて遺伝子同定する方法論を樹立する必要がありました。そこで死にゆく細胞の遺伝子同定を可能とするために、CRISPR Cas9 sgRNAライブラリー注4)を用いた新しいスクリーニング法(リバイバルスクリーニング)を樹立しました。リバイバルスクリーニングでは以下の手法で遺伝子同定を行います。①レンチウイルスに組み込まれたsgRNAライブラリーを標的細胞に導入後、アポトーシス刺激を行い、PSを露出しない細胞を、FACS注5)を用いて回収します。②回収した細胞は、アポトーシス刺激下にあるため死滅します。そこで、回収した細胞からゲノムDNAを精製し(DNAの断片化を防ぐためにDNase注6)は欠損させてある)、sgRNAのターゲット配列を含む領域をPCR法にて増幅し、そのPCR産物をレンチウイルスベクターに挿入し、濃縮されたsgRNAライブラリーを構築します。③構築されたsgRNAライブラリーを用いてレンチウイルスを作製し、再び標的細胞に感染させスクリーニングを行います。④このプロセスを複数回行うことで、PS露出に関わる活性化因子が高濃度で濃縮され、次世代DNAシークエンス解析により、スクランブラーゼXkr4の活性化因子を同定することができました(図3)。驚くことに、その活性化因子はDNA修復に関与するXRCC4でした。XRCC4は通常は核内に存在し、損傷を受けたDNAの修復に関わっています。しかしながら細胞が死んだ時には、XRCC4がカスパーゼによって切断されることで、折りたたみ構造をとることのない、天然非変性領域から成るC末端側が細胞質に放出され、細胞膜中のXkr4に直接結合することでXkr4を活性化させることが分かりました(図4)。核内タンパク質の断片が細胞膜タンパク質を活性化させるという報告はなく、これまでの常識を超えた発見です。これにより、脳内において不要な細胞が除去される分子機構の一端が解明されました。
今後の展開
今後、様々な生体システムでリバイバルスクリーニングを行うことにより、新しい因子の発見が期待されます。また、スクランブラーゼXkr4は神経細胞において不要な細胞だけでなく、生きた細胞の不要なシナプスなど、細胞の一部分を除去するのにも関わると考えられます。今後、神経変性疾患や精神疾患がどのように発症するのか、そのメカニズムを理解するうえにおいても、本研究成果は重要な位置を占めると考えられます。
用語解説
- ※1 貪食
- マクロファージなどの食細胞は死んだ細胞を食べることで炎症を防ぐと考えられている。
- ※2 スクランブラーゼ
- 細胞膜のリン脂質を双方向に輸送することで、“死んだ目印”となるホスファチジルセリン(PS)を細胞表面に露出する。
- ※3 DNA修復
- 損傷したDNAを元通りに修復すること。
- ※4 CRISPR Cas9 sgRNA library
- 細菌由来のタンパク質(Cas9)を用いて、標的とするDNAに相補的なRNA(sgRNA)を用いてDNAの編集を行う技術。対象とする細胞の遺伝子を網羅的に編集できるsgRNA群(sgRNA library)を用いることで遺伝子の発見を可能とする。
- ※5 FACS
- 細胞にレーザーをあてることで、細胞と反応する蛍光プローブを検出し、目的の細胞を分取することができる解析機器。
- ※6 DNase
- 細胞が死ぬ時に活性化してDNAを分解する酵素。
研究プロジェクトについて
日本医療研究開発機構(AMED)革新的先端研究開発支援事業(PRIME)「画期的医薬品等の創出をめざす脂質の生理活性と機能の解明」研究開発領域(研究開発総括:横山信治)における研究開発課題「細胞膜における脂質動態の制御機構の解明とその応用」(研究開発代表者:鈴木淳)、FORCEにおける研究開発課題「細胞膜脂質動態の異常による神経疾患発症の理解並びにその治療戦略の提案」(研究開発代表者:鈴木淳)
若手A(KAKENHI 15H05651),
新学術領域研究“Scrap & Build”(KAKENHI 16H06456),
Joint Usage and Joint Research Programs of the Institute of Advanced Medical Sciences of Tokushima University,
WPI-iCeMS,
Takeda Science Foundation
Ono Foundation to J.S.,
MEXT Grants-in-Aid for Scientific Research (C) (KAKENHI 20K06486) to M.M.
論文情報
- 論文タイトル
- “Caspase cleavage releases a nuclear protein fragment that stimulates phospholipid scrambling at the plasma membrane”
- (参考訳:核内タンパク質の断片が細胞膜のリン脂質スクランブルを活性化する)
- 著者
- Masahiro Maruoka, Panpan Zhang, Hiromi Mori, Eiichi Imanishi, Daniel Packwood, Hiroshi Harada, Hidetaka Kosako, Jun Suzuki
- 掲載誌
- Mol Cell
- DOI
- 10.1016/j.molcel.2021.02.025
お問い合わせ先
研究内容について
鈴木淳(スズキ・ジュン)
京都大学アイセムス(物質-細胞統合システム拠点)教授
お問い合わせ | 京都大学アイセムス
京都大学アイセムスについて
遠山真理(トオヤマ・マリ)高宮泉水(タカミヤ・イズミ)
京都大学アイセムス(物質-細胞統合システム拠点)
パブリックエンゲージメントユニット
お問い合わせ | 京都大学アイセムス
AMEDの事業について
国立研究開発法人日本医療研究開発機構
シーズ開発・研究基盤事業部 革新的先端研究開発課
E-mail:kenkyuk-ask“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
関連リンク
掲載日 令和3年3月31日
最終更新日 令和3年3月31日


