成果情報 mRNAの安定性は遺伝暗号コドンの組み合わせによって変化する。その原因は「リボソームの減速」
成果情報
京都産業大学
理化学研究所
日本医療研究開発機構
研究概要
タンパク質は、DNAから転写されたメッセンジャーRNA(mRNA)をもとに、その上の塩基の並び(コドン[注1])をリボソーム[注2]が3塩基ずつ解読し、アミノ酸を順番に連結することで合成されます。mRNAが安定だと多くのタンパク質が継続して合成されます。mRNAは解読が終わると分解されますが、分解のタイミングが早いと合成されるタンパク質の量は少なくなります。mRNAの安定性がうまく調整されていれば、必要な量のタンパク質が正しいタイミングで合成されるのですが、この安定性の調節が破綻すると、がん、炎症性疾患、神経変性疾患など、様々な病気の原因ともなります。リボソームが解読するmRNAのコドンの組み合わせが、mRNAの安定性(寿命の長さ)を左右する要因の1つであることはこれまでにも知られていましたが、その基本的な仕組みはよくわかっていませんでした。
今回私たちは、新たにPACE法という分析手法を開発し、小型熱帯魚ゼブラフィッシュの胚において、コドンがmRNAの安定性にどのような影響を与えるかを測定しました。そして、「コドンを解読するリボソームの減速が、mRNAの分解を引き起こす」ことを突き止めました。また、この「リボソームの減速」は一時的なものであり、タンパク質合成時のトラブルによって引き起こされる「リボソームの異常な渋滞」とは異なる現象であることも示しました。
これらの結果から、「mRNAからタンパク質が作られる際のリボソームの移動のスピードが、mRNA自身の寿命を決めている」ことを明らかにしました。
本研究の成果は、私たちの体の中でmRNAとタンパク質の量が調節される際の基本原理の理解を飛躍的に高めることになります。また、本研究で得られた知見は、mRNAワクチンの最適化や、生体内でのコントロール技術の創出にも貢献できると考えます。
背景
生物の細胞の中では、様々なタンパク質が、細胞の機能や生存に必要な様々な反応を進行させています。私たちの体を作る約300種類の細胞を正常に機能させるには、2万種類以上ともいわれるタンパク質から必要な組み合わせを見つけ、それを適切な量だけ作らなければなりません。
タンパク質は、ゲノムDNAの遺伝子の情報(遺伝情報)に基づいて、アミノ酸分子を1つずつ連結させることで合成されます。このときDNAの遺伝情報は、まず、メッセンジャーRNA(mRNA)に写し取られます(転写)。次に、タンパク質合成装置であるリボソームが、mRNAの塩基を端から3塩基ずつ解読し、対応するアミノ酸を順番に連結することで、様々なタンパク質を合成します(翻訳)。リボソームによって解読される3つ一組の塩基の組み合わせはコドンと呼ばれ、tRNAという分子がそれぞれのコドンに対応するアミノ酸をリボソームへと運びます。コドンとアミノ酸の対応関係は、遺伝暗号と呼ばれています。
タンパク質の設計図であるDNAは壊れにくく非常に安定ですが、一時的なコピーであるmRNAは、分解されやすいものからされにくいものまで、その安定性(寿命の長さ)は様々です。mRNAの分解されやすさ/されにくさは、タンパク質がどれくらいの期間、どれくらいの量が作られるのかによって大きく変わってきます。例えば炎症反応に関わるタンパク質は炎症応答の初期にのみ必要なので、そのmRNAは数分で分解されてしまいます。一方、細胞の中で常に必要なタンパク質のmRNAは、数時間経っても分解されないものもあります。このようなmRNAの安定性を決める情報もまた、mRNAの塩基配列として書き込まれているのです。
これまで、mRNAの安定性を決める配列情報のほとんどは、mRNAのうちリボソームによって翻訳されない部分(UTR)にあるという考え方が主流だったのですが、数年前に、私たちを含め複数の研究グループが、様々な生物において、「遺伝暗号であるコドンの組み合わせが、mRNAの安定性を決定している」ことを発見しました(引用:Mishima and Tomari Mol Cell. 2016など)。
ただし、タンパク質のアミノ酸の並び順を決めるための暗号であるはずのコドンの組み合わせが、mRNAの安定性の情報として読み出される仕組みは、これまでよくわかっていませんでした。今回、私たちは、モデル生物である淡水性の小型熱帯魚ゼブラフィッシュを使った実験により、コドンの組み合わせがどのようにしてmRNAの安定性の違いを生じさせるのか、その仕組みを明らかにしました。
研究方法と成果
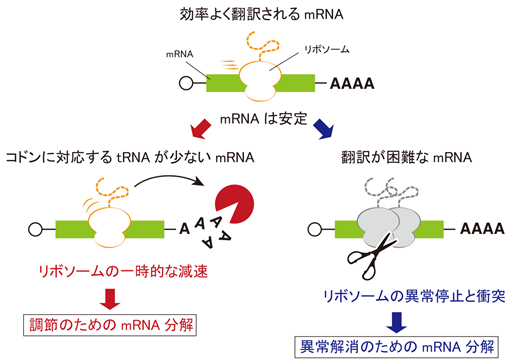
今回私たちは、コドンがmRNAの安定性に与える影響を解析するために、PACE法(Parallel Analysis of Codon Effects)という実験方法を開発しました。PACE法では、さまざまなコドンを持つmRNAを試験管内で合成し、それぞれのmRNAの安定性を同時並行で解析することで、これまで測定が困難であった「各コドンがどれくらいmRNAの分解を促進するのか」を数値化することができるようになりました。このPACE法をゼブラフィッシュの受精卵に導入して解析を行ったところ、mRNAを分解する効果を持つコドンには、2つの共通した特徴があることがわかりました。一つは「コドンに対応するtRNAの量が少ないこと」、もう一つは「リボソームがコドンを解読するのにより長い時間がかかること」です。tRNAの量が少ないとリボソームがコドンを解読する際に減速することはこれまでにも知られていましたが、この特徴がmRNAの分解につながっていることは実証されていませんでした。そこで、実験的に特定のtRNAの量を減らしたところ、対応するコドンがmRNAの分解を促進する効果を獲得することが確認できました。
ところで、リボソームがmRNAを翻訳する際のスピードは、tRNAの量以外にも様々な要因によって変化します。特にリボソームが翻訳しにくいコドンやアミノ酸の並びに差し掛かると、リボソームの異常停止と衝突が起こり、‘交通渋滞’のような状況に陥ります。細胞には、このような交通渋滞を解消するためのリボソーム品質管理[注3]の機構が備わっており、その品質管理の際にもmRNAが分解されることが知られていました。私たちは、mRNAの分解を促進するコドンの上でのリボソームの減速は一時的なもので、交通渋滞には至っていないことを確認しました。つまり、コドンの組み合わせによるmRNAの安定性の変化は、リボソーム品質管理のためのmRNAの分解とは別の、独立した仕組みによってもたらされているわけです。これらの結果は、細胞の中ではリボソームがmRNAを翻訳する際のさまざまな動きが個別に感知されていて、特にリボソームの一時的な減速が、mRNAの安定性に大きな影響を与えていることを示しています。
今後の展開
PACE法を用い、様々な生物種や細胞種においてコドンがmRNAの安定性にどのような影響を与えるのかを調べることで、遺伝子の配列情報からmRNAの安定性をより高精度に予測することが可能になると考えられます。この情報は、mRNAから合成されるタンパク質の量と機能時期の推定や、突然変異によりコドンに変化が起きた場合の影響の予測に利用できると期待されます。また、mRNAワクチンのような人工mRNAが、細胞の中で安定かつ持続的にタンパク質を作り出せるように設計する技術の開発にも貢献できると期待しています。
用語・事項の解説
- [注1]コドン
- リボソームが解読する3塩基一組の組み合わせ。64個のコドンによって、20種類のアミノ酸と翻訳の終結が指定されている。
- [注2]リボソーム
- 細菌からヒトまで、すべての生物が持つタンパク質合成装置。mRNAの配列(コドンの並び)にしたがって、アミノ酸を1つずつ連結し、タンパク質を合成する。
- [注3]リボソーム品質管理
- mRNAを翻訳している途中で起こったリボソームの異常停止を解消するしくみ。翻訳を強制終了させてリボソームを再利用し、mRNAとタンパク質を分解することで、翻訳中のリボソーム、mRNA、タンパク質の異常を解消する。
論文情報
- 論文タイトル
- Ribosome slowdown triggers codon-mediated mRNA decay independently of ribosome quality control(リボソームの減速はリボソーム品質管理と独立にコドンによるmRNA分解を引き起こす)
- 掲載誌
- 欧州分子生物学機関誌「The EMBO Journal」(オンライン版)
- 掲載日
- 2022年1月18日(火)(日本時間)20時00分
- 著者
- 三嶋 雄一郎1, 2, 3、Peixun Han4、石橋 幸大3、木村 成介3、岩崎 信太郎4
- (1筆頭著者、2責任著者、3京都産業大学、4理化学研究所)
- DOI
- 10.15252/embj.2021109256
本研究への支援
この研究は、科研費の基盤研究 (B) (JP18H02370, JP21H02513) 、若手研究(A)(JP17H04998)、挑戦的研究(萌芽)(JP19K22406)、新学術領域研究 (JP17H05593, JP17H05662)、学術変革領域研究(B)(JP20H05784)、日本医療研究開発機構(AMED)・革新的先端研究開発支援事業(PRIME)「健康・医療の向上に向けた早期ライフステージにおける生命現象の解明」研究開発領域,同(AMED-CREST)「プロテオスタシスの理解と革新的医療の創出」研究開発領域、文部科学省の私立大学戦略的研究基盤形成支援事業(S1511023)、稲盛財団の助成を受けて実施されました。
お問い合わせ
研究に関する問い合わせ
京都産業大学 生命科学部 准教授 三嶋 雄一郎(みしま ゆういちろう)
TEL:075-705-1943
E-mail:mishima"AT" cc.kyoto-su.ac.jp
AMED事業に関する問い合わせ
日本医療研究開発機構(AMED)
シーズ開発・研究基盤事業部 革新的先端研究開発課
〒100-0004 東京都千代田区大手町1-7-1 読売新聞ビル21階
TEL:03-6870-2224
E-mail:kenkyuk-ask"AT" amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
関連リンク
掲載日 令和4年1月31日
最終更新日 令和4年1月31日


