2019年度 研究事業成果集 核酸医薬の創薬に資する霊長類のオミックスデータベースD3Gの公開
 ヒト・非ヒト霊長類のゲノム、遺伝子等の高品質データを提供
ヒト・非ヒト霊長類のゲノム、遺伝子等の高品質データを提供
理化学研究所予防医療・診断技術開発プログラムの河合純副プログラムディレクターらの共同研究グループは、ヒト、カニクイザル、マーモセットの霊長類のゲノムと遺伝子モデル、遺伝子発現の情報を含むオミックス情報の統合データベース「D3G(Database for Drug Development based on Genome and RNA)」を公開しました。本データベースを利用することで、創薬に携わる研究者の業務が大きく推進され、創薬における新たなイノベーションにつながると期待できます。
取り組み
アンチセンス医薬やsiRNA医薬に代表される核酸医薬は、種特異性が高いため、ヒトと遺伝的な類似度が高い非ヒト霊長類を用いた評価が有用と考えられます。しかし、これまで非ヒト霊長類の遺伝子情報の整備は限定的であるとともに体系的に整理されておらず、創薬のボトルネックになっていました。
今回、本研究グループは、創薬研究で用いる非ヒト霊長類モデル動物のカニクイザルとマーモセットのゲノムDNAとRNAを独自技術を駆使して解析し、高品質で総合的なデータセットを構築しました。
本データセットは全ゲノムDNA配列、遺伝子モデル、毒性評価試験の対象とされる全組織の遺伝子発現情報を含み、塩基配列検索ツールとともにデータベースD3Gとして公開されています。
本データベースのα版(限定版)を、2019年夏より研究や創薬を目的とした組織や団体に公開し、2020年3月には最新情報を掲載した正式版を一般公開しました(図1)。本データベースのデータは、高速塩基配列検索ソフトウェア「GGGenomeパッケージ版(創薬パック)」(株式会社レトリバ)にも収録されています(図2)。
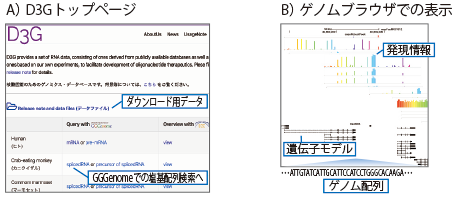

成果
本研究グループは、研究コミュニティが必要とするデータベースを開発するために、多くの製薬企業、規制関係者、学会の協力を得て、求められるデータコンテンツを明らかにしました。発育段階や産地にも考慮してサンプリングし、カニクイザルとコモンマーモセットに関するDNAゲノムとRNAのデータを取得しました。取得したデータセットには、高品質のゲノム配列と毒性評価試験の対象とされる全組織の遺伝子発現情報という際立った特徴があります。
ゲノム配列については、染色体レベルでのゲノム配列構築に成功しています。この新しいゲノム配列に含まれるギャップ領域(塩基配列が読み取れていない領域)の個数を、これまで用いられてきたカニクイザルとコモンマーモセットのゲノム配列に比べて百分の一に減らすことができました。これは、ヒトやマウスのゲノム配列に迫る高品質のゲノム配列です。
また、カニクイザル、コモンマーモセットのそれぞれから、毒性評価試験対象となる約50種類の臓器について、分解を最小限に抑えてRNAを採取し、RNA-seqとCAGEのデータを取得することで(カニクイザルで18,333個、マーモセットで18,629個)、ヒトやマウスと同等の完成度の遺伝子モデルを構築できました(表1)。これらにより、核酸医薬の開発に重要な遺伝子塩基配列の情報と組織ごとの遺伝子発現の情報を提供しています。
| ヒト | カニクイザル | コモンマーモセット | マウス | |
|---|---|---|---|---|
| ゲノム アセンブル内のギャップ数+ |
349 (GRCh38) |
605* | 1,847$ |
191
(GRCm38)
|
| 遺伝子モデル タンパク質コード遺伝子座の数 |
19,423 (RefSeq release109) |
18,333* | 18,629* | 20,634 (RefSeq release109) |
+:塩基配列がよみとれていない領域,*:本研究で取得したデータから構築した配列情報,$:共同研究を通じて得られたデータから構築した配列情報
展望
ヒト・非ヒト霊長類およびマウスについて精度の高いオミックス情報を整理、統合し提供しているD3Gは、核酸医薬のオフターゲット効果の予測・評価などに有用とされ、国立医薬品食品衛生研究所などとの共同研究として『Genes to Cells』に発表した「RNA分解型アンチセンスのオフターゲット効果の評価法」に関する論文*1においても、D3Gのヒトpre-mRNAのデータセットが活用されています。また、2020年3月に薬事承認された日本新薬株式会社のデュシェンヌ型筋ジストロフィー核酸医薬ビルトラルセンの申請*2においてもD3Gが使用されました。
本成果は、医科学や生物学などの基礎研究の基盤データを提供するにとどまらず、創薬の研究開発の加速などに大きく貢献するものです。今後、データを充実させるとともに、疾患や遺伝子機能に関する情報を連携させることが期待されます。
- *1
- Tokuyuki Yoshida, Yuki Naito, Hidenori Yasuhara, Kiyomi Sasaki, Hideya Kawaji, Jun Kawai, Mikihiko Naito, Haruhiro Okuda, Satoshi Obika, Takao Inoue.Evaluation of off-target effects of gapmer antisense oligonucleotides using human cells. Genes to Cells, Vol.24, No.12, 827-835(2019)
- *2
- 審議結果報告書 2020年3月6日 厚生労働省 医薬・生活衛生局医薬品審査管理課
最終更新日 令和3年8月13日


