プレスリリース 体の一日を刻むタイマー機構を発見―遺伝子導入マウス個体高速作成法を用いた概日時計周期長制御の解明―
プレスリリース
国立研究開発法人理化学研究所
国立大学法人東京大学
国立研究開発法人日本医療研究開発機構
要旨
理化学研究所(理研)生命システム研究センター合成生物学研究グループの上田泰己グループディレクター、大出晃士客員研究員、鵜飼英樹上級研究員、洲﨑悦生客員研究員らの共同研究グループ※は、さまざまな遺伝子改変マウスを並列的に作製する手法を確立し、「クリプトクロム1(CRY1)」と呼ばれるタンパク質の特定の領域がリン酸化によって制御されることが「概日時計」の周期の長さ(周期長)を決めるために重要であることを発見しました。
私たちが夜眠りにつき朝目覚めるように、地球上の生物の多くは24時間周期のリズムを持って行動しています。こうした行動リズムは、概日時計と呼ばれる全身の細胞が持つ時計機能によって生み出されます。しかし、周期長がなぜ24時間なのかは、不明な点が多く残されています。
今回、共同研究グループはさまざまに機能を変化させたタンパク質を持つ遺伝子改変マウスを効率よく作製する新しい手法「遺伝子導入マウス個体高速作製法」を確立しました。そしてこの手法により、概日時計機能を失ったマウスに、さまざまに機能を変化させたタンパク質をコードする遺伝子を導入し、概日時計機能を補完することが可能となりました。その結果、概日時計を動かすために重要な働きをするCRY1タンパク質の特定の領域がタイマーのように働き、マウスの周期長を決定していることを発見しました。このタイマー領域は、生体内でタンパク質機能の制御によく用いられるリン酸化と呼ばれる化学修飾(リン酸化修飾)を受けていることから、タンパク質内のリン酸化修飾が巧みに時間を数えることで、正確に24時間の周期を刻む可能性が強く示唆されました。
今後、このタイマー領域のリン酸化を薬物で制御することができれば、概日時計の周期長を効率的にコントロールし、概日リズム睡眠障害[1]等の効果的な治療につながると期待できます。
また、遺伝子導入マウス個体高速作製法を用いると、全身の細胞が遺伝子改変されたマウスをこれまで1年以上かけて作製していたのを数カ月で作製できようになります。そのため、この手法により遺伝子改変マウスを用いる多くの研究が加速すると期待できます。
本成果は米国の科学雑誌『Molecular Cell』(1月5日号)に掲載されるのに先立ち、オンライン版(12月22日付け:日本時間12月23日)に掲載されます。
本研究は、国立研究開発法人日本医療研究開発機構 革新的先端研究開発支援事業(AMED-CREST)の研究開発領域「生体恒常性維持・変容・破綻機構のネットワーク的理解に基づく最適医療実現のための技術創出」(研究開発総括:永井 良三)における研究開発課題「睡眠・覚醒リズムをモデルとした生体の一日の動的恒常性の解明」(研究開発代表者:上田 泰己)の一環で行われました。なお、本研究開発領域は、平成27年4月の日本医療研究開発機構の発足に伴い、国立研究開発法人科学技術振興機構(JST)より移管されています。
背景
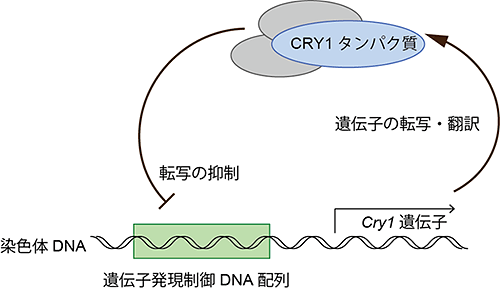 図1.CRY1タンパク質による概日時計の発振機構
図1.CRY1タンパク質による概日時計の発振機構
クリプトクロム1(CRY1)タンパク質は、遺伝子発現制御DNA配列の下流にある遺伝子の転写を抑制する。Cry1遺伝子もこの制御下にあるため、CRY1タンパク質は自身の生産を抑制する。この抑制によって、CRY1タンパク質が多いときにはCRY1タンパク質の生産が抑えられ、やがてCRY1タンパク質が減少すると、CRY1タンパク質が再び生産されるプロセスが周期的に繰り返される。
そこで共同研究グループは、CRY1タンパク質による概日時計の周期長の制御を包括的に明らかにするために、➀CRY1タンパク質のどの部分が周期長を調整するのかをCRY1タンパク質全域から網羅的に特定し、②特定した領域が周期長の制御に本当に重要なのかをマウスの行動リズムの変化として厳密に検証する計画を立てました。
特に、共同研究グループは➁を実施するために、さまざまに機能を変化させたCRY1タンパク質を持つ遺伝子改変マウスを効率よく作製する手法の確立を目指しました。
研究手法と成果
1)CRY1タンパク質による概日時計の周期長制御に関する解析
概日時計の周期長の制御に重要なCRY1タンパク質の領域を調べるにあたって、共同研究グループはリン酸化修飾に着目しました。リン酸化は、細胞内でタンパク質の機能制御に用いられる化学修飾です。まず、質量分析計[2]を用いてCRY1タンパク質を構成するアミノ酸配列の中でリン酸化修飾を受けている場所(リン酸化アミノ酸残基)を網羅的に調べました。次に、これらのアミノ酸残基が一つずつ異なるアミノ酸残基に変化するように設計した「変異Cry1遺伝子シリーズ」を作製しました。特に、リン酸化されたアミノ酸残基をリン酸化されていない状態によく似た異なるアミノ酸残基に置換(疑似非リン酸化変異[3])したり、逆にリン酸化された状態に似たアミノ酸残基に置換(疑似リン酸化変異[3])したりすることで、それぞれ対応するアミノ酸残基がリン酸化されていないときやリン酸化されているときのCRY1タンパク質の性質を調べました。
次に、これらの変異Cry1遺伝子シリーズが概日時計の周期長をどのように制御するか調べました。そのために、Cry遺伝子群(Cry1遺伝子とCry2遺伝子)を欠損させ概日時計機能を失った細胞(Cry1-/-:Cry2-/-細胞[4])に、プラスミドDNA[5]で野生型Cry1遺伝子または変異Cry1遺伝子シリーズを導入し、概日時計機能を補完する実験を行いました(Cry1遺伝子機能補完実験[6]※注1。この実験では、野生型Cry1遺伝子だけを導入し細胞の概日時計機能を補完したときと、変異Cry1遺伝子シリーズだけを導入し補完したときとで、周期長がそれぞれどのように変化するかが分かります。
実験の結果、野生型よりも長いあるいは短い周期の概日時計機能を補完する変異Cry1遺伝子を多数発見しました(図2)。特に、243番目のアミノ酸残基とその近くにあるアミノ酸残基はリン酸化されることで、周期長を大幅に短くすること、その影響は複数のリン酸化が同時に生じると加算的に増加し、いわばタイマーのように働きうることが強く示唆されました。
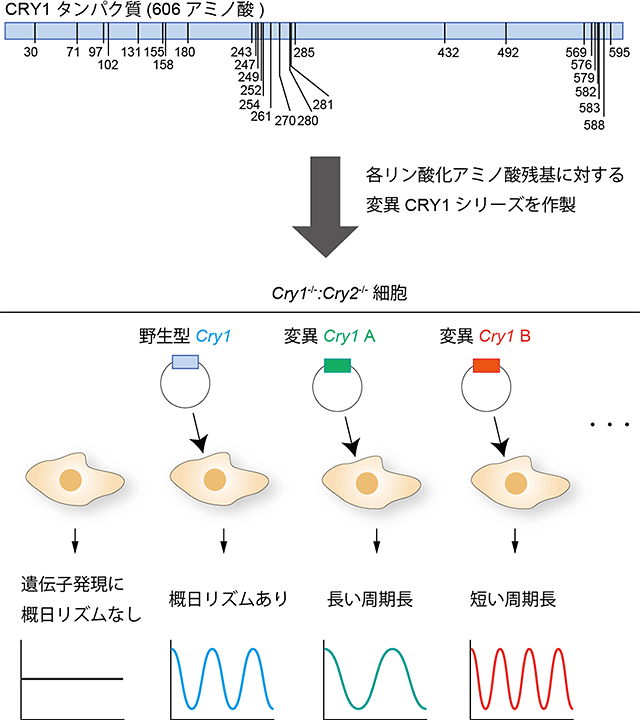 図2.培養細胞のCry1遺伝子機能補完実験
図2.培養細胞のCry1遺伝子機能補完実験
CRY1タンパク質は606個のアミノ酸配列からなる。このうち、図に示した番号のアミノ酸残基がリン酸化されることが分かった。「野生型Cry1遺伝子」または、それぞれのアミノ酸残基が異なるアミノ酸に置き換わるように設計した「変異Cry1遺伝子シリーズ」を、概日時計機能を失ったCry1-/-:Cry2-/-細胞にプラスミドDNAで導入することで、概日時計機能を補完した。その結果、変異Cry1遺伝子シリーズの中に、野生型Cry1遺伝子よりも長いあるいは短い周期の概日時計機能を補完する変異Cry1遺伝子を多数発見した。
- ※注1 2011年1月14日プレスリリース「夕方の遺伝子発現の仕組みと役割を初めて解明」 ※理化学研究所ホームページより
2)遺伝子導入マウス個体高速作製法
共同研究グループは、概日時計の周期長に影響を与える変異Cry1遺伝子が、マウスの行動リズムに与える影響を調べるために、胚性幹細胞(ES細胞)から作製した個体を利用してCry1遺伝子の機能補完実験を行う新たな手法を確立しました。
この手法ではまず、Cry遺伝子群を欠損したマウス(Cry1-/-:Cry2-/-マウス)から、体のさまざまな種類の細胞に分化できるES細胞を作製します。次に、このES細胞(Cry1-/-:Cry2-/- ES細胞)のROSA26と呼ばれる染色体上の領域にCry1遺伝子を導入(遺伝子ノックイン[7])することで、欠損したCry遺伝子群の働きを補完します。そして、Cry1遺伝子をノックインしたCry1-/-:Cry2-/-ES細胞を、さまざまな細胞に分化する能力を維持できる条件で培養します。その後、培養したCry1-/-:Cry2-/-ES細胞を発生初期のマウスの胚に移植して生体マウス(ESマウス[8])を作製します。
2010年、理研生命システム研究センター生体モデル開発ユニットの清成寛ユニットリーダーらは、こうして得られるESマウスは体を構成する細胞がほぼ全て移植したES細胞に由来することを報告しました※注2。このようにESマウスは全身のほとんどの細胞が遺伝子改変後のES細胞からできているため、交配の必要がなく、従来のノックインマウス作製の手順に比べて、より高速にかつ並列的に行うことができるという利点があります。今回確立した手法は、Cry1遺伝子のノックイン以外にも、多様な遺伝子についてノックインマウスを作製することに広く応用できる手法です(遺伝子導入マウス個体高速作製法)。
※注2 Kiyonari et al., Genesis, 48, 317-327 (2010). Three inhibitors of FGF receptor, ERK, and GSK3 establishes germline-competent embryonic stem cells of C57BL/6N mouse strain with high efficiency and stability
3)CRY1タンパク質による概日時計の周期長の制御に関する解析
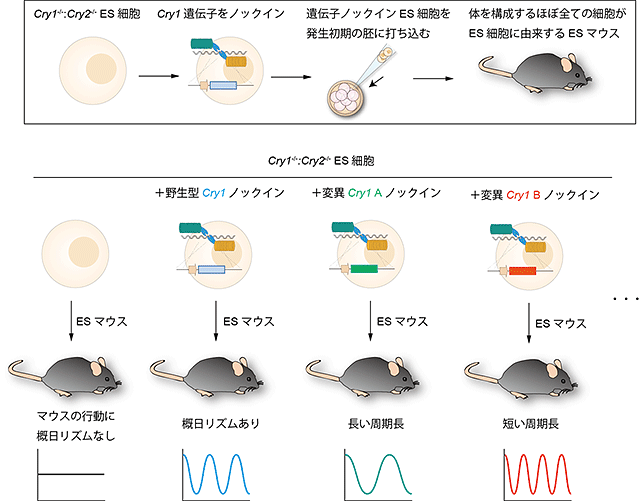 図3.遺伝子導入マウス個体高速作製法で作製したESマウスの行動リズム測定
図3.遺伝子導入マウス個体高速作製法で作製したESマウスの行動リズム測定
上: Cry1-/-:Cry2-/-マウスから作製したES細胞(Cry1-/-:Cry2-/- ES細胞)に、Cry1遺伝子をノックインする。次に、このES細胞(Cry1-/-:Cry2-/-ES細胞)を発生初期のマウス胚に注入する。すると成長したマウスは、全身の細胞がこのES細胞に由来するESマウスとなる。
下: ノックインするCry1遺伝子を野生型や、変異Cry1遺伝子シリーズに変えることで、さまざまな種類のESマウスを作製し、行動リズムの周期長を測定した。
また、一部の変異Cry1遺伝子を導入し細胞の概日時計機能を補完したESマウスは安定した概日リズムを示さず、これらの変異CRY1タンパク質は「PER2」と呼ばれるタンパク質との相互作用が低下していました。PER2タンパク質は生化学的な役割に不明な点が多いタンパク質であり、この発見はPER2タンパク質の役割を深く理解する手掛かりになる可能性があります。
4)CRY1タンパク質の特定の領域は、CRY1タンパク質の分解活性の変化とは異なる仕組みで概日時計の周期長を制御
これまで細胞内でCRY1タンパク質が分解される速度の変化が、概日時計の周期長を制御するために重要であると考えられてきました。そこで、共同研究グループは今回発見したマウスの行動の概日リズムを制御する変異CRY1タンパク質のリン酸化アミノ酸残基が、分解速度に影響を与えるかを調べました。その結果、変異CRY1タンパク質の中には、分解速度が変化するものが複数ある一方で、概日時計の周期長に大きく影響を与える243番目やその周辺のリン酸化アミノ残基に変異を導入した変異CRY1タンパク質の多くは、分解速度に大きな影響がみられませんでした。
さらに、AID法(オーキシン誘導デグロン法[9])を用いて、野生型CRY1タンパク質を分解したところ、周期長が変化しました。これらの結果から、いくつかの変異CRY1タンパク質は、従来の考え通り分解速度の変化が周期長を制御していると考えられます。一方、周期長を大きく変えた変異CRY1タンパク質は、分解速度の変化と別の原因で、周期長を制御していると考えられます。
続いて、CRY1タンパク質の分解速度に影響を与えないが、周期長を大幅に変えるリン酸化アミノ酸残基の特徴を調べました。その結果、これらのアミノ残基はP-loop、C-lidと呼ばれるCRY1タンパク質の領域を取り囲む場所に集中していることが分かりました(図4)。特に、P-loop周辺には243番目のリン酸化アミノ酸残基を含めて、加算的に周期長を変化させると考えられるアミノ酸残基が存在します。
これまでの研究から、これらの領域はCRY1タンパク質の中でも比較的構造が変化しやすいことが予想されています。リン酸化はこれらの領域の構造変化を誘導し、周期長を変化させる可能性があります。実際に、共同研究グループが、予想される構造変化に影響を与えるようにP-loopやC-lidのアミノ酸残基を変化させたところ、概日時計の周期長に大きな影響がある一方で、CRY1タンパク質の分解速度は影響されないことが分かりました。
これらの結果から、P-loop、C-lid 領域がタイマーとして働き、領域内のリン酸化が加算的に概日時計の周期長を制御している可能性が示されました。
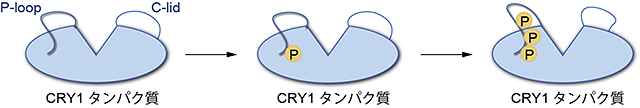
図4.CRY1タンパク質のタイマー領域の複数のリン酸化による概日時計制御モデル
CRY1タンパク質にあるP-loop、C-lidと呼ばれるタイマー領域周辺のリン酸化は、特に概日時計の周期長を変化させる。特にP-loop周辺に存在するリン酸化アミノ酸残基への複数のリン酸化の影響は、加算的に周期長を変化させる可能性が強く示唆された。
今後の期待
本研究で確立した遺伝子導入マウス個体高速作製法を用いると、全身の細胞が遺伝子改変されたマウスを1年以上かけて作製していたのを数カ月で作製できようになります。これにより、概日時計研究以外にも、睡眠のような個体全体が関わる生理現象と、特定の性質を持ったタンパク質との関係を調べる上で、重要な技術になると期待できます。
また、これまで知られていたCRY1タンパク質の分解速度とは異なるメカニズムで周期長が著しく変化し、それに関わるCRY1タンパク質のタイマー領域(P-loop、C-lid)のリン酸化修飾の周期長制御における意義を明らかにしたことで、概日時計研究の新たな展開につながると考えられます。
さらに、本研究で示したような、特定の領域周辺に生じる複数のリン酸化によって概日時計の周期長が制御される例は、哺乳類や他の生物種の異なる概日時計関連のタンパク質にも発見されています。時間情報を制御するために複数のリン酸化を用いる戦略は、生物種を超えて保存されているのかもしれません。
今回発見した概日時計の周期長を強力に制御するリン酸化残基を薬物で制御することができれば、周期長を効率的に制御し、概日リズム睡眠障害等の効果的な治療につながると期待できます。
論文情報
- タイトル:
- Knockout-Rescue Embryonic Stem Cell-Derived Mouse Reveals Circadian-Period Control by Quality and Quantity of CRY1
- 著者名:
- Koji L. Ode, Hideki Ukai, Etsuo A. Susaki, Ryohei Narumi, Katsuhiko Matsumoto, Junko Hara, Naoshi Koide, Takaya Abe, Masato T. Kanemaki, Hiroshi Kiyonari, Hiroki R. Ueda
- 雑誌:
- Molecular Cell
- DOI:
- 10.1016/j.molcel.2016.11.022
補足説明
- [1] 概日リズム睡眠障害
- 概日リズムが昼夜の24時間リズムと合わなくなることで起きる睡眠障害。例えば、概日リズムが24時間よりも長いと、夜になっても寝付けず、朝起きることができないなどが起きやすい。
- [2] 質量分析計
- 分子の質量を精密に測定する分析装置。ペプチドにリン酸化修飾があると、リン酸基に応じた質量変化が生じるため、質量分析計を用いてペプチドがリン酸化されていることを検出できる。
- [3] 疑似非リン酸化変異、疑似リン酸化変異
- リン酸基は細胞内でマイナスに荷電しているため、これらの残基をマイナス電荷を持つアスパラギン酸に変異させると、リン酸化された状態を疑似的に作ることができる(疑似リン酸化変異)。また、これらのアミノ残基をリン酸化を受けず電荷を持たないアラニンなどのアミノ酸に変異させると、リン酸化されていない状態を疑似的に作ることができる(疑似非リン酸化変異)。本研究では、セリン・スレオニン・チロシンへのリン酸化に着目した。
- [4] Cry1-/-:Cry2-/-細胞
- ゲノムDNA上の二つのCry遺伝子(Cry1とCry2)を欠損させ機能しないようにした細胞。二つのうち一方だけを欠損しても、細胞の概日時計は発振する機能を保つ。そのため、二つのCry遺伝子を機能しないようにすることで細胞の概日時計を止め、Cry1遺伝子の発現メカニズムやCRY1タンパク質の機能解析を可能とした細胞。
- [5] プラスミドDNA
- 宿主であるバクテリアのゲノムDNAとは物理的に独立して自律複製し、安定に存在できる遺伝因子。ベクター(運び屋)として利用される。
- [6] Cry1遺伝子機能補完実験
- 細胞の概日リズムは、概日時計のある時刻に活性が上昇する遺伝子発現制御DNA配列の下流でホタル発光遺伝子であるルシフェラーゼを発現させることで、発光量変動として観測できる。Cry1-/-:Cry2-/-細胞は発光量の周期的な変動はみられない。この細胞に、適切な遺伝子発現制御DNA配列の下流でCry1遺伝子を発現するプラスミドDNAを導入すると、発光量の周期的な変動がみられるようになる。
- [7] 遺伝子ノックイン
- 細胞は傷ついたDNAを修復する手段として相同組換え修復経路を持っているが、これを利用して高い効率で遺伝子を導入する手法。本研究では、TALENと呼ばれる特定のDNA配列を切断する働きを持つタンパク質を、ROSA26領域を切断するように設計して用いることにより、この領域を狙って傷をつけ、相同組換え修復を誘発することで、高い効率で遺伝子ノックインさせる工夫を行った。
- [8] ESマウス
- 2010年に理研生命システム研究センター生体モデル開発ユニットの清成寛ユニットリーダーらによって報告された発生工学の手法を用いて作製したマウス。この手法では、ES細胞を3種類の異なる細胞内シグナルの阻害剤存在下で培養し、さまざまな細胞種に分化する能力を高いレベルで維持させる。このES細胞を発生初期の8細胞期の胚に移植すると、体のほぼ全ての細胞が移植されたES細胞に由来するESマウスを得られる。ES細胞に遺伝子組換えを行い、マウスでの表現型を解析する際、通常のES細胞から作製したマウスは、体を構成する一部の細胞がES細胞由来となるキメラマウスとなるため、全身の細胞がESマウスと同じ遺伝子型を持つマウスを得るにはマウスを交配し、その子孫を実験に用いる必要があった。
- [9] AID法(オーキシン誘導デグロン法)
- 2009年に国立遺伝学研究所の鐘巻将人教授ら(当時 大阪大学)によって開発された方法。植物では植物ホルモンであるオーキシン依存的にタンパク質を分解するシステムが存在する。このタンパク質分解経路の一部が動物細胞にも共通することを利用し、動物細胞にオーキシン応答に必要なタンパク質を導入することで、動物細胞内でオーキシン依存的なタンパク質分解を誘導する。本研究では、CRY1タンパク質がオーキシン依存的に分解されるように実験系を設計した。AIDはauxin-inducible degronの略。
お問い合わせ先
発表者※研究内容については発表者にお問い合わせください
理化学研究所 生命システム研究センター 合成生物学研究グループ
グループディレクター 上田 泰己(うえだ ひろき)
(東京大学 大学院医学系研究科機能生物学専攻 教授)
客員研究員 大出 晃士(おおで こうじ)
(東京大学 大学院医学系研究科機能生物学専攻 助教)
上級研究員 鵜飼 英樹(うかい ひでき)
客員研究員 洲﨑 悦生(すさき えつお)
(東京大学 大学院医学系研究科機能生物学専攻 助教)
TEL:03-5841-3415(上田) FAX:03-5841-3418(上田)
E-mail: uedah-tky“AT”umin.ac.jp(上田)
機関窓口
理化学研究所 広報室 報道担当
TEL:048-467-9272 FAX:048-462-4715
E-mail:ex-press“AT”riken.jp
東京大学 大学院医学系研究科 総務係
TEL:03-5841-3304 FAX:03-5841-8585
E-mail:ishomu“AT”m.u-tokyo.ac.jp
日本医療研究開発機構(AMED)戦略推進部医薬品研究課
TEL:03-6870-2219
E-mail:20-DDLSG-16“AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
※共同研究グループ
- 理化学研究所
- 生命システム研究センター 合成生物学研究グループ
グループディレクター 上田 泰己(うえだ ひろき)ライフサイエンス技術基盤研究センター 生体モデル開発ユニット
(東京大学 大学院医学系研究科機能生物学専攻 教授)
客員研究員 大出 晃士(おおで こうじ)
(東京大学 大学院医学系研究科機能生物学専攻 助教)
上級研究員 鵜飼 英樹(うかい ひでき)
客員研究員 洲﨑 悦生(すさき えつお)
(東京大学 大学院医学系研究科機能生物学専攻 助教)
元テクニカルスタッフ 鳴海 良平(なるみ りょうへい)
特別研究員 松本 桂彦(まつもと かつひこ)
元テクニカルスタッフ 原 淳子(はら じゅんこ)
元テクニカルスタッフ 小出 直史(こいで なおし)ユニットリーダー 清成 寛(きよなり ひろし)生体ゲノム工学研究チーム専門職研究員 阿部 高也(あべ たかや) - 国立遺伝学研究所
- 分子細胞工学研究部門 鐘巻研究室
教授 鐘巻 将人(かねまき まさと)
掲載日 平成28年12月23日
最終更新日 平成28年12月23日


