プレスリリース 細胞表面の情報センサーの基本原理を解明―センサータンパク質に作用するくすりの開発に貢献―
プレスリリース
東北大学
日本医療研究開発機構
発表のポイント
- くすりの主要な標的となる、細胞表面のセンサータンパク質が細胞に情報を伝える仕組みを明らかにした。
- 機械学習を用いて、遺伝子情報(アミノ酸配列)から細胞表面センサーの機能を予測する手法を開発した。
- 細胞に特定の情報を入力することのできる人工センサーを創製した。
- 情報伝達の知見を利用することで、細胞表面センサーに作用する新たな疾患治療薬や副作用を抑えたくすりの開発につながる。
概要
東北大学大学院薬学研究科の井上飛鳥准教授、青木淳賢教授とドイツハイデルベルク大学のRussell博士らの研究グループは、くすりの主要な作用標的である細胞表面に存在するタンパク質群の情報伝達様式を解明しました。私たちの体にはこのタンパク質群が約280種類存在し、ホルモンに応答する情報伝達センサーとして個々の細胞に備わっています。このセンサータンパク質の機能異常は数々の疾患を引き起こします。くすりは異常となったセンサータンパク質に結合して、その機能を正常化することで疾患を治す働きがあります。一方で、このセンサータンパク質が細胞に伝える情報の種類は多様であり、その一部は副作用に関わることが知られているものの、全容は分かっていませんでした。今回、研究グループは約150種類にも及ぶセンサータンパク質群の情報伝達様式を明らかにしました。得られた実験データを基に、センサータンパク質のアミノ酸配列から情報伝達様式を高精度に予測するアルゴリズムを開発し、残りの約130種類のセンサーの情報伝達様式をスコア化するとともに、特定の情報を入力することのできる人工センサーを作製しました。この研究成果は、薬効と副作用の分子機序の解明に貢献するとともに、未だ治療薬のない疾患に対するくすりの開発を加速することが期待されます。
この研究成果は、米国科学誌Cellのオンライン版に2019年5月31日(日本時間)に掲載されます。
本研究は、日本医療研究開発機構革新的先端研究開発支援事業ソロタイプ(PRIME)『画期的医薬品等の創出をめざす脂質の生理活性と機能の解明』研究開発領域(研究開発総括:横山信治)における研究開発課題「リガンドが不要な革新的GPCRツールを用いた脂質関連オーファン受容体の機能解明(研究代表者:井上飛鳥)」、インキュベートタイプ(LEAP)における研究開発課題「リゾリン脂質メディエーター研究の医療応用(研究代表者:青木淳賢)、文部科学省科学研究費補助金等の支援を受けて行われたものです。
詳細な説明
背景
私たちの体を構成する細胞は常時適切な情報を受け取り、体の恒常性を支えています。ホルモンはこのような細胞外の情報伝達分子であり、細胞の表面に存在する受容体と呼ばれるホルモンセンサーに結合することで、細胞の内部へ信号を伝えます。受容体の中でもGタンパク質共役型受容体(GPCR、*用語1)と呼ばれる一群(ヒトには約280種類存在することがゲノム解読から判明しています)が重要な役割を担うことがわかっています。GPCRは特定のホルモン様分子(リガンドと呼ばれます)と結合すると三量体Gタンパク質(以下、Gタンパク質、Gs、Gi、Gq、G12の4グループに分類されます。*用語2)と呼ばれる細胞内タンパク質に情報を受け渡します。GPCRごとに固有のGタンパク質の情報伝達パターンが存在し、このパターンに従い細胞の振る舞いが決まります。また、GPCRが正常に機能しなくなると細胞の振る舞いに異常をきたし、数多くの疾患の原因となることが知れています。GPCRに結合しその機能を元に戻す作用を有する分子は疾患治療薬となります。実際にGPCRに作用するくすりは、市販薬の約3割と数多く存在することが知られており、現在でも多くの研究者や企業がGPCRを標的とした創薬に取り組んでいます。全てのGPCRについて、ホルモン様分子が結合した際に生じるGタンパク質の結合パターンを明らかにすることができれば、疾患機序の分子レベルでの解明や新たなくすりの開発につながります。
GPCRにホルモン様分子が結合すると、構造変化(活性化、*用語3)を起こし、Gタンパク質が結合できるようになります(この結合は共役と呼ばれ、GPCRの名称の由来となっています)。GPCRと結合したGタンパク質は、細胞内に存在する核酸分子(GTP)を取り込み、このGTP結合型(活性型)のGタンパク質が別のタンパク質と結合することで細胞内の情報伝達が進行します(情報伝達を信号の流れとして捉え、後半を下流と呼びます)。GPCRに結合するGタンパク質を調べるには、その結合を直接測定するのが困難なため、通常は下流の細胞応答を調べます。例えば、Gsの下流ではサイクリックAMP濃度の上昇、Gqの下流では細胞内へのカルシウムイオン流入が生じます。しかし、異なる細胞応答現象を比較してその強弱は判断できないことから、GPCRにどのGタンパク質がどの程度結合するかを測定することは困難でした。加えて、4つのGタンパク質グループ(Gs、Gi、Gq、G12)にはそれぞれ複数の種類が存在していますが、1つのグループは似通った細胞応答を引き起こすため、個々のGタンパク質の結合を測定することはできませんでした。
本研究では、これまでに本研究者らが開発したGPCR活性化の測定方法を改良することで、GPCRと個別のGタンパク質との結合を測定する方法を確立するとともに、多種類のGPCRについて個別の結合を測定することに成功し、GPCRに作用する創薬に有用な情報を構築しました。
研究手法と成果
特定のGPCRとGタンパク質の結合を解析する手法
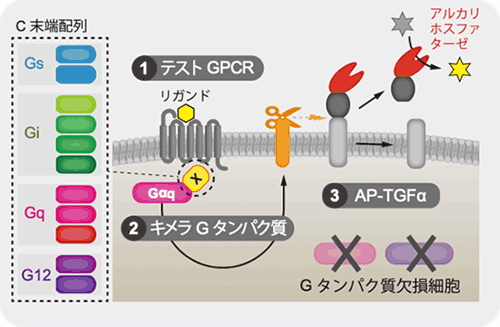
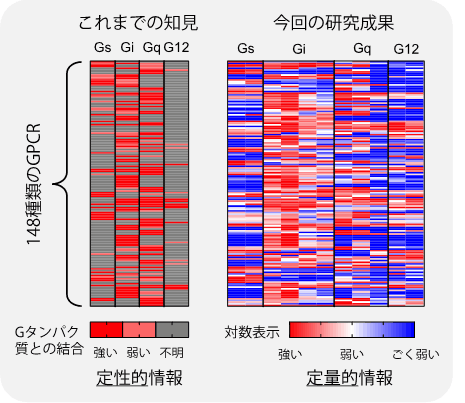
本研究者らは、以前の研究成果(Nat Methods. 9, 1021-1029 (2012))において、トランスフォーミンング増殖因子アルファ(TGFα)のタンパク質切断を利用したGPCR活性化(=Gタンパク質の結合と活性化に伴う情報伝達)の測定手法(TGFα切断アッセイ、*用語4)を開発しました。今回、この実験手法をGタンパク質欠損細胞(*用語5)と人工改変Gタンパク質(キメラGタンパク質*用語6)と組み合わせて実施することで、ホルモン様分子がGPCRに結合した際のキメラGタンパク質との結合を個々に測定する技術を構築しました(図1)。つまり、細胞に導入したキメラGタンパク質のみがGPCRと結合しTGFα切断を引き起こす状況を作り出すことで、GPCRとGタンパク質の結合を特異的に測定する手法です。この手法を用いて、148種類のGPCRについて全てのキメラGタンパク質との結合を測定しました。この結果、既存の手法では成し得なかった、詳細なGPCRとGタンパク質の相互作用パターンを明らかにしました(図2)。特に、G12のグループに関して多くのGPCRが結合することがわかりました。
Gタンパク質の選択性に重要なアミノ酸残基の解析
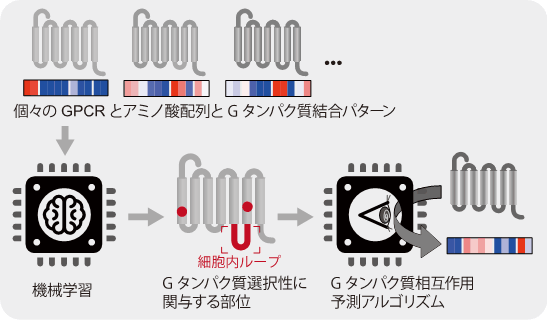
次に、実験的に得られたGPCRとGタンパク質の結合パターンから、Gタンパク質の選択性に関わるGPCRのアミノ酸配列を明らかにすべく、生物情報学(*用語7)の手法を用いた解析を行いました。その結果、細胞膜貫通領域や細胞内ループに各Gタンパク質を識別することに関与するアミノ酸残基が存在することがわかりました。これまでの解析から、一部のGPCRについてGタンパク質と直接相互作用するアミノ酸残基の重要性は知られていましたが、意外なことに、今回の生物情報学解析で見出されたGタンパク質の選択性に関与するGPCRのアミノ酸残基の多くは、やや離れた位置(細胞外のリガンド結合の近く)に存在することがわかりました(図3)。すなわち、GPCRに共通なGタンパク質の選択性を決める因子は、Gタンパク質との相互作用面に存在するのではなく、この面の構造を変化させる蝶番(ちょうつがい)に相当する位置にあると言えます。
Gタンパク質結合の予測アルゴリズムの開発
続いて、上記のGタンパク質の選択性に関与するアミノ酸配列の情報を基に、機械学習を用いてGタンパク質結合を予測するアルゴリズム(Predictor)を開発しました。公開データベース(IUPHAR/Guide to PHARMACOLOGY)に登録されたGタンパク質との結合情報を指標に、今回のアルゴリズムと既存のアルゴリズム(PredCouple)を比較したところ、4種類のGタンパク質グループのいずれについても、今回のアルゴリズムの方が、既存よりも同等かより精度が高くGタンパク質結合を予測できることがわかりました。さらに、このアルゴリズムを用いて、解析が進んでいない61種類のGPCR(結合分子が未同定、情報伝達が不明)のGタンパク質結合スコアを算出すると、よく解析されているGPCRと比べてG12と結合する割合が高いことが予測されました。
Gタンパク質結合の選択性の改変
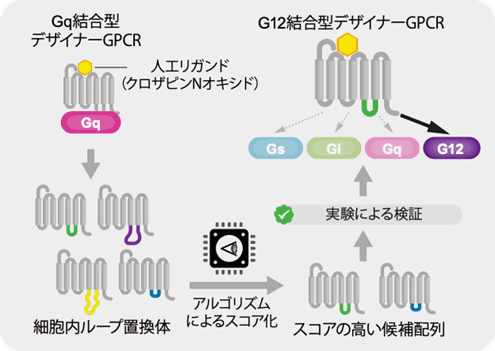
最後に、予測アルゴリズムを用いてGタンパク質の選択性を改変することを試みました。そのモデルとして、デザイナーGPCR(*用語8)と呼ばれる受容体を用いました。デザイナーGPCRは、生体内のホルモン様分子には応答せず、特定の合成分子のみにより機能がオンとなる人工改変受容体です(図4)。予測アルゴリズムを用いて、Gqと結合するデザイナーGPCR(M3D)を改変することで、G12と結合するデザイナーGPCRを作製することを試みました。M3Dのリガンド結合部位は変えずに細胞内ループを置換した配列を288種類設計し、コンピューターでスコア化しました。このうち、上位26種類について実際に受容体遺伝子を合成して実験を行なったところ、4種類の人工受容体がG12と結合し、情報伝達を引き起こすことがわかりました。このうち、G12シグナル活性の高かった2種類について詳細に解析したところ、Gqを含めた他のGタンパク質とは結合せず、G12のみに作用することがわかりました(図4)。
応用範囲の広いGPCR解析技術の構築
本研究では、上記のキメラGタンパク質を利用したTGFα切断アッセイ以外にも複数のGPCRの解析技術を開発し、上記の解析結果の検証を行いました。例えば、Gs欠損細胞とGs骨格のキメラGタンパク質の組み合わせは、サイクリックAMPの増加を指標とすることで、TGFα切断アッセイと同様に、GPCRとGタンパク質の相互作用を検出できます。Gq欠損細胞を用いたTGFα切断アッセイは、G12の情報伝達を特異的に検出できることから、G12シグナル解析への応用が見込まれます。また、補酵素断片法NanoBiTシステム(プロメガ社)を利用した原理の異なる実験手法、具体的にはGタンパク質の活性化レベルを発光を指標に測定する手法(NanoBiT-Gタンパク質アッセイ)、低分子Gタンパク質RhoAの活性化測定手法(NanoBiT-RhoAアッセイ)、細胞内のイノシトール三リン酸(IP3)の測定手法(NanoBiT-IP3センサー)を作成しました。
このうち、特にNanoBiT-Gタンパク質アッセイは、各々のGタンパク質について活性型の量を簡便に直接測定できる手法であり、今後、くすりの性状解析を始めとして様々なGPCR解析に利用されることが期待されます。
今後の期待
細胞の主要な情報センサーであるGPCRの情報伝達様式の基本原理を解明したことで、分子レベルでの病気の理解やくすりの開発の効率化に貢献することが期待されます。例えば、今回解明したGPCRとGタンパク質の結合情報を基に、疾患時に大きく変化するGタンパク質情報伝達経路を見出すことができます。つまり、情報伝達の上流でGタンパク質の情報伝達を正常化させるGPCRを絞り込むのに有用であり、このGPCRが疾患治療薬の標的候補となり得ることを示しています。今回の研究で、多くのGPCRがG12と結合することが判明したことから、開発したG12結合デザイナーGPCRと合わせて解析を進めることで、G12の情報伝達に基づいた新しいくすりの創製の道筋が拓けるものと考えられます。
用語説明
- 1.Gタンパク質共役型受容体(GPCR)
- 細胞膜に存在する膜型タンパク質であり、細胞外のホルモン様分子と結合し、細胞内へ情報を伝えるセンサー。細胞膜を貫通するヘリックスを7個有する特徴的な構造から、7回膜貫通型受容体とも呼ばれる。ヒトゲノムに存在する約800種類のうち、クラスA(ロドプシンファミリー)と呼ばれるグループに分類される約280種類が古典的なホルモン受容体として研究が進んでいる。これまでに開発されたくすりの約3割がGPCRと結合して作用を発揮することから、現在も創薬開発における重要な標的として研究されている。GPCRはG-Protein-Coupled Receptorの略。
- 2.三量体Gタンパク質
- GPCRと直接結合(共役と呼ばれます)し、GPCRのホルモン結合状態を読み取り、細胞内の別の分子へと情報を伝達するタンパク質。Gα、Gαβ、Gαγの3つのサブユニットのタンパク質複合体である。三量体Gタンパク質の性質はGαサブユニットの種類で決まる。ヒトゲノムには16種類のGαサブユニットが存在し、アミノ酸配列と情報伝達の種類から4種類(Gs、Gi、Gq、G12)に分類される。Gαサブユニットは結合する核酸によりオンオフが切り替わり、グアノシン三リン酸(GTP)と結合する時にGβγサブユニットから離れて活性型の構造を取る。GTP結合Gαサブユニットは自身のリン酸基を分解する酵素活性(GTP分解活性、Gタンパク質の名称の由来)によりグアノシン二リン酸(GDP)結合型となり、Gβγと会合して元の状態に戻る。
- 3.タンパク質の活性化
- タンパク質の機能がオンになっている構造。タンパク質の運動とは必ずしも関係ない。GPCRの場合、ホルモン様分子との結合型、三量体Gタンパク質の場合、GTP結合型が該当する。活性型GPCRはGタンパク質を活性型へと変化させ、活性型Gタンパク質は別のタンパク質と相互作用することでその機能を制御する。この一連の流れを細胞の情報伝達(シグナル伝達)と呼び、情報の始まり側と終わり側をそれぞれ上流と下流と表現する。
- 4.TGFα切断アッセイ
- 膜タンパク質TGFαの切断反応を利用したGタンパク質の活性化測定手法。GqとG12の情報伝達の下流で膜型タンパク質分解酵素の機能がオンとなり、TGFαの細胞外部位を切断する。TGFαにアルカリホスファターゼ(AP)を融合した改変体タンパク質(AP-TGFα)を用いることで、TGFαの切断量を細胞培養液のAPの比色反応として簡便に定量できる。今回の研究では、AP-TGFα、評価対象のGPCR、キメラGタンパク質(11種類のうち1種類)をリポフェクション法によりGタンパク質欠損細胞に発現させ、このGPCRに対する結合分子(リガンド)を添加した際のAP-TGFαの切断量をGPCRとGタンパク質の結合活性として評価した。Transforming Growth Factor-Alpha(トランスフォーミンング増殖因子アルファ)の略。
- 5.Gタンパク質欠損細胞
- Gαサブユニットの遺伝子に変異を導入してその機能を欠失させた細胞。今回の研究では、本研究者らが以前ゲノム編集技術(CRISPR-Cas9システム)を用いてGqとG12のグループを欠損させたHEK293細胞(ヒト胎児腎臓細胞由来、リポフェクション法による遺伝子導入が容易なことで知られる)を使用した。このGタンパク質欠損細胞はTGFα切断を誘導するGタンパク質が発現していないため、遺伝子導入したキメラGタンパク質のTGFα切断活性を特異的に測定できる。
- 6.キメラGタンパク質
- Gαサブユニットのカルボキシル末端(C末)側を改変したタンパク質。GαサブユニットのC末側がGPCRとの主要な相互作用部位であることから、キメラGタンパク質は伝達する情報の種類は変えずに、GPCRの活性型構造を読み取ることができる。16種類のGαサブユニットのうち、重複を除いてC末側の6アミノ酸は11種類存在する。今回の研究ではGαqサブユニットのC末側の6アミノ酸残基を他のGαサブユニットの配列と置換した11種類の改変体(およびネガティブコントロールとしてC末端を欠失した改変体1種類)を用いた。
- 7.生物情報学(バイオインフォマティクス)
- 生物学の実験データを情報科学の手法を利用して解析する手法。今回の研究の場合、GPCRのアミノ酸配列(平均300アミノ酸程度)と結合するGタンパク質(11種類)を解析した148種類のGPCRについて比較し、Gタンパク質の選択性と相関するアミノ酸配列の特徴を機械学習の手法を用いて抽出した。
- 8.デザイナーGPCR
- リガンド結合部位が改変され、特定の合成分子で受容体機能が制御されるGPCR。本来のホルモン様分子は結合しない。ムスカリン性アセチルコリン受容体の改変体とクロザピンNオキシドの組み合わせが汎用されている。今回の研究ではGqと結合するM3Dの細胞内ループを改変し、クロザピンNオキシドとの親和性は保ったままG12の活性化を誘導する人工受容体を作製した。別名のDREADD(ドレッド)はDesigner Receptors Exclusively Activated by Designer Drugsの略。
論文目録
- 英語タイトル:
- Illuminating G-protein-coupling selectivity of GPCRs.
- 日本語タイトル訳:
- Gタンパク質共役型受容体のシグナル選択性の解明
- 著者:
- Asuka Inoue(井上 飛鳥、責任著者), Francesco Raimondi, Francois Marie Ngako Kadji, Gurdeep Singh, Takayuki Kishi, Akiharu Uwamizu, Yuki Ono, Yuji Shinjo, Satoru Ishida, Nadia Arang, Kouki Kawakami, J. Silvio Gutkind, Junken Aoki(青木 淳賢), Robert B. Russell
- 掲載誌:
- Cell
- 掲載日時
- 電子版:2019年5月30日(現地時間)、冊子版:2019年6月13日(現地時間)、177号、1-15ページ
- DOI:
- 10.1016/j.cell.2019.04.044
お問い合わせ先
東北大学大学院薬学研究科
担当 井上 飛鳥(准教授)
TEL:022-795-6861/022-795-4528
E-mail:iaska”AT”tohoku.ac.jp
AMED事業に関するお問い合わせ
国立研究開発法人日本医療研究開発機構(AMED)
基盤研究事業部 研究企画課
革新的先端研究開発支援事業
TEL:03-6870-2224
E-mail:kenkyuk-ask”AT”amed.go.jp
※E-mailは上記アドレス“AT”の部分を@に変えてください。
掲載日 令和元年5月31日
最終更新日 令和元年5月31日


