東北メディカル・メガバンク計画で「日本人基準ゲノム配列」初版JG1を公開
 従来よりも精密かつ正確な日本人のゲノム解析が可能に
従来よりも精密かつ正確な日本人のゲノム解析が可能に
東北大学東北メディカル・メガバンク機構(ToMMo)は、日本人ゲノム解析のひな型となる「日本人基準ゲノム配列」の初版JG1を作成し、日本人多層オミックス参照パネル(jMorp)で完全公開しました。このJG1は日本人3名のゲノム配列を基に複数の方法を組み合わせて精密に決定されました。JG1を用いることで、高精度の全ゲノム解析が可能となり、これまで未特定だった遺伝性疾患の原因究明やがんを引き起こす遺伝子の同定などが期待されます。
取り組み
ゲノム医療の推進には正確なゲノム解析が重要であり、基準となるゲノム配列に対して、調べたい個人との差を検出する方法がよく用いられています。これまで基準となるゲノム配列は、ヨーロッパ系とアフリカ系の集団を祖先に持つゲノムを基に作成された「国際基準ゲノム」が主流でしたが、対象が日本人の場合には解析困難な箇所(難読領域)があり、本来あるべき違いが検出されなかったり、誤検出などの問題点が指摘されたりしていました。また、遺伝性が強く疑われる疾患の約半数は、原因となる遺伝子の同定ができておらず、これは全ゲノム解析の精度が十分でないことが要因の一つと考えられます。
このような問題点の克服を目的として、難読領域を含めた日本人の全ゲノム配列を解読するとともに、国際基準ゲノムの影響や個人によって異なる希少な配列バリエーションの影響を排除するため、日本人3名に由来するゲノム配列を基に複数の方法を組み合わせて高精度に統合した「日本人基準ゲノム配列」の構築に挑戦しました(図1)。

成果
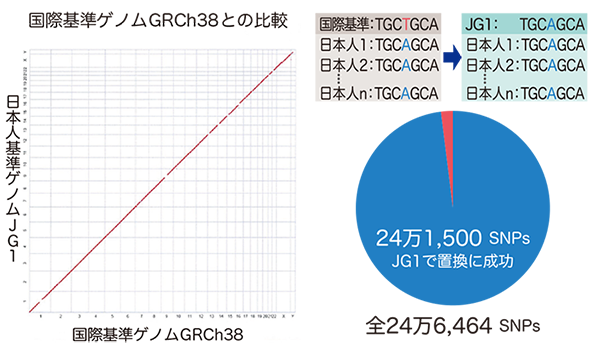
出身地域が異なる3名の日本人男性の全ゲノム配列の決定を行いました。ゲノムを1万塩基対程度の比較的長い単位(長鎖)で解読できる長鎖リードと、解読したゲノム配列間を結合するためのオプティカルマッピング*1という2種類のゲノム解読技術を用い、さらに短鎖技術による次世代シーケンシング解析で解読エラーを除き、高精度な配列スキャフォールド*2を作成しました。その上で、メイトペアライブラリー技術*3を利用して3者間で解読できなかった領域を補い合って、3者の配列を統合しました。その後、3者で異なる部分は多数派の配列を採用し、一個人に由来する配列バリエーションを取り除いて最後に、既知の遺伝地図等の情報を用いて配列スキャフォールドとヒト染色体との対応付けを行い、日本人基準ゲノム配列JG1を完成させました。このようにして構築したJG1が、上述の本来あるべき違いが検出されなかったり、誤検出といった問題点の解決に寄与するかを「国際基準ゲノムGRCh38」とJG1で比較検討しました。その結果、国際基準ゲノム配列には日本人集団には見られない配列バリエーションが24万6,464カ所で観察されたのに対し、JG1ではそのうちの24万1,500カ所を日本人の大多数に見られるアレルに置換していることが分かりました(図2)。これは、JG1を利用すれば日本人の次世代シーケンシング解析において短鎖リードの当てはめや配列バリエーション検出エラーを劇的に減らし、より精密で正確なゲノム診断を可能にすることを示唆するものです。一塩基多型に限らずより大きな構造バリアントについても、多数派への置換を行っており、国際基準ゲノム配列を用いた解析に存在するバイアスを取り除くことが期待されます。
- *1オプティカルマッピング:
- 特定の塩基配列に施した蛍光ラベル間の距離を観測し、物理地図を作成することで、シーケンサーで解読した配列を整列させる技術。
- *2配列スキャフォールド:
- 物理地図や遺伝地図等により隣接することが判明した配列間を、未解読を意味する連続するNNN...Nで連結した塩基配列。
- *3メイトペアライブラリー技術:
- 本技術と短鎖技術との併用により数千から一万数千塩基のDNA断片の両端の塩基配列情報を正確な向き情報とともに取得することができる。
展望
JG1は、現在主流の一つである短鎖技術による次世代シーケンシング解析を、日本人を対象として行う場合に、国際基準ゲノム配列の代わりに利用することができます。具体的には、①小児の希少疾患の原因解明、②日本人全ゲノムリファレンスパネルの再構築、③がんゲノムエクソーム解析への応用、④ヒトゲノム突然変異率の推定への利用が考えられます。
最終更新日 令和2年6月23日


